par Patrick PLA, Université Paris-Saclay
La découverte dans les années 1970 des enzymes de restriction, des enzymes bactériennes qui peuvent couper l’ADN au niveau de séquences spécifiques, a ouvert la voie à toute une série de manipulations du matériel génétique. Les chercheurs disposent maintenant d’une palette d’outils à la fois pour séquencer un fragment d’ADN (voire tout un génome) et pour étudier l’expression des gènes mais aussi pour la modifier.
- Le séquençage
- L’analyse GWAS
- L’hybridation in situ
- RT-qPCR
- RNAseq
- Les gènes rapporteurs
- La fixation des facteurs de transcription à l’ADN
- Détection des modifications épigénétiques de la chromatine
- Détection des structures chromatiniennes
- Etude de la traduction
- Modifications du génotype
- Les outils bioinformatiques
Le séquençage
Les techniques classiques de séquençage reposent sur l’utilisation de didésoxyribonucléotides (et non des désoxyribonucléotides comme on trouve habituellement dans l’ADN). Ces nucléotides particuliers se retrouvent forcément en bout de la chaîne nucléotidique car aucun autre nucléotide ne peut s’ajouter à leur extrémité 3′. En faisant synthétiser par une ADN polymérase un brin d’ADN à partir d’une matrice dont on veut connaitre la séquence et en utilisant un didésoxyribonucléotide précis (par exemple celui dont la base est la guanine), on sait que toutes les chaînes vont se terminer par G. Selon leur taille, ainsi que leurs tailles relatives par rapport à des synthèses où on aura utilisé des nucléotides modifiés avec d’autres bases azotées, on peut reconstituer la séquence d’un fragment d’ADN. Initialement, on utilisait des nucléotides radioactifs. Maintenant on utilise des nucléotides attachés à un fluorophore, une couleur différente pour chaque didésoxyribonucléotide avec une base différente. On peut alors lire la séquence après séparation des brins de différentes longueurs en mesurant la fluorescence des 4 canaux possibles correspondant à A, T, C et G.

Les méthodes de séquençage modernes dites à haut débit lisent la fluorescence en cours de synthèse et non plus une fois la synthèse des brins effectués ce qui permet de gagner en rapidité. Le séquençage classique est réalisé sur des fragments de 500 à 600 bases, alors qu’il n’y a pas véritablement de limite pour les méthodes modernes. Le coût du séquençage a chuté drastiquement en conséquence (le séquençage d’un génôme humain en 2001 coûtait 100 millions de $; aujourd’hui, il coûte 700 $). Le séquençage d’un génome humain qui était une véritable aventure scientifique à la fin des années 1990/début des années 2000 est maintenant quelque chose de « banal ».
L’analyse GWAS
Il s’agit d’une étude d’association sur tout le génome (pangénomique) (ou GWAS en anglais pour genome-wide association study) entre des variations génétiques chez de multiples individus et des traits phénotypiques (typiquement des maladies génétiques). Cela permet de localiser dans le génome des allèles associés à un phénotype donné. Généralement, les marqueurs de variations utilisés sont les SNP (ou single nucleotide polymorphism), qui représentent 90% des variations génétiques humaines et qui sont bien répartis dans tout le génome (même si certaines régions sont plus riches en SNP, notamment les séquences non codantes).
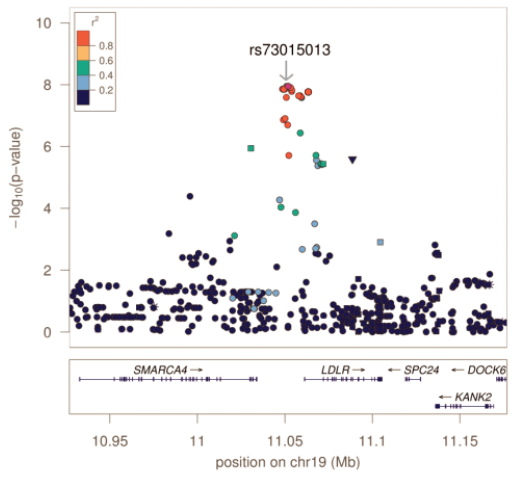
Analyse des SNP associés à des taux de cholestérol LDL élevés, dans la région du génome où se trouve le gène codant le récepteur aux LDL (LDLR). Chaque point est un SNP. En ordonnées, est indiqué le niveau d’association entre les SNP et un taux de cholestérol élevé. La couleur des points reflète le déséquilibre de liaison des SNP dans la population considérée : plus la couleur est chaude, plus la probabilité que les allèles ségrégent avec le SNP considéré est élevée.
La mutation sur le SNP rs73015013 n’est peut-être pas la cause directe
de l’augmentation du taux de cholestérol, mais elle est située dans une
région qui ségrége fréquemment avec ce trait phénotypique. Source : https://journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.1002198
L’hybridation in situ
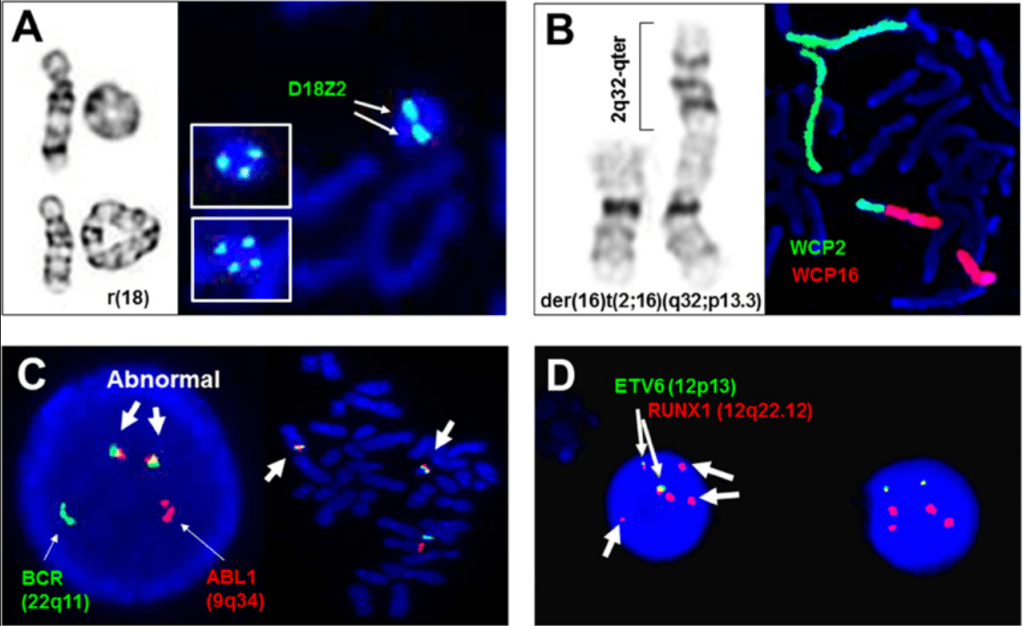
Le terme hybridation in situ regroupe deux techniques qui se ressemblent par certains aspects mais qui ont un objectif différent. L’une permet de détecter des gènes dans un génome et notamment sur des chromosomes et l’autre permet de détecter des ARNm ou des microARN sur des embryons entiers ou des coupes histologiques.
Pour détecter une séquence d’ADN génomique par fluorescence in situ sur les chromosomes (FISH), on utilise une sonde fluorescente d’ADN simple brin avec la séquence complémentaire de celle qui nous intéresse. On peut faire deux FISH en parallèle avec des fluorophores différents. L’ADN est coloré grâce au DAPI.
Pour détecter des ARNm, le principe de complémentarité des bases des acides nucléiques est utilisé. On tire parti de la nature simple brin de l’ARNm et on introduit une séquence complémentaire à l’ARNm cible avec des modifications qui permettent sa visualisation. D’abord cette sonde doit être synthétisée in vitro. Il s’agit d’une molécule d’ARN antisens dont la longueur peut varier entre 200 pb à 2000 pb. La synthèse de cet ARN se fait en présence d’une UTP (uridine triphosphate) conjuguée à la digoxygénine, un composé fabriqué par un groupe particulier de végétaux (les Digitales) et introuvable dans les cellules animales.
Un ARN sens (séquence identique à l’ARNm cible) est aussi produit et sert de témoin négatif (il ne doit pas reconnaître lui-même) et permettra d’évaluer la part du marquage qui correspond à du bruit de fond. L’embryon est fixé (typiquement avec du 4% paraformaldéhyde) puis est perméabilisé par des solvants lipidiques (Tween-20 ou Triton X100) et des protéinases (protéinase K) afin que la sonde puisse entrer et sortir de ses cellules. Une fois dans les cellules, l’hybridation se produit entre l’ARN anti-sens de la sonde et l’ARNm ciblé. Pour visualiser les cellules dans laquelle l’hybridation s’est produite, les chercheurs utilisent un anticorps qui reconnaît spécifiquement la digoxygénine. Cet anticorps a été artificiellement conjugué à une enzyme, telle que la phosphatase alcaline. Après incubation dans l’anticorps et des lavages répétés pour éliminer tous les anticorps non liés, l’embryon est baigné dans une solution contenant un substrat pour l’enzyme (traditionnellement un mélange NTB/BCIP pour la phosphatase alcaline) qui donne un produit coloré en bleu-violet. En incubant de plus en plus longtemps les tissus avec les substrats, on peut révéler des expressions de plus en plus faibles. Cependant, cette méthode de détection n’est pas quantitative.

D’après https://mmegias.webs.uvigo.es/02-english/6-tecnicas/5-hibridacion.php

Des méthodes plus modernes utilisent des sondes fluorescentes complémentaires des ARNm ce qui permet de facilement observer l’expression de plusieurs ARNm à la fois et ces sondes peuvent aussi être utilisées en culture cellulaire (Young et al., 2020).
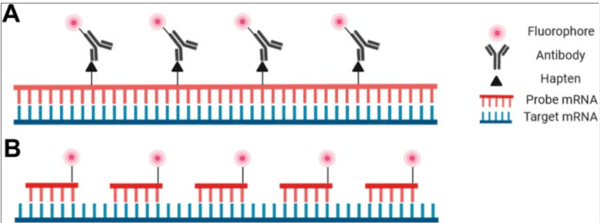
Des nouvelles techniques comme le RNAscope permettent d’améliorer encore la sensibilité de l’hybridation in situ grâce à un système d’amplification : voir cette vidéo de présentation :
Voici les différentes étapes :
- Hybridation des sondes cibles (Z-probes) :
Deux demi-sondes (appelées ZZ pairs) s’hybrident à l’ARN cible en tandem. Ce n’est que si les deux parties d’une paire ZZ se fixent l’une à côté de l’autre sur l’ARN cible que la structure est reconnue comme valide, ce qui augmente la spécificité.
- Fixation du “Pre-amplificateur” :
Chaque paire de sondes ZZ crée un site d’ancrage pour une molécule de pré-amplification (pre-AMP), qui ne peut se lier que si les deux moitiés de la sonde sont présentes et correctement positionnées.
- Fixation des “Amplificateurs” :
Chaque molécule de pré-amplificateur permet la fixation de plusieurs molécules d’amplificateurs. Ces molécules contiennent à leur tour plusieurs sites de liaison pour les molécules de détection (soit fluorophores TSA, soit substrats enzymatiques).
- Fixation des sondes de détection (fluorophore ou HRP) :
Enfin, les amplificateurs se lient aux sondes de détection, souvent via un système Tyramide Signal Amplification (TSA) si l’on utilise des fluorophores. Une molécule d’ARN peut ainsi être marquée par des dizaines à des centaines de fluorophores, rendant le signal visible même si l’ARN est faiblement exprimé.
RT-qPCR
Un exemple d’étude avec une RT-qPCR :
Pour faire une RT-qPCR, il faut d’abord extraire les ARN des cellules ou des embryons.
Exemple de protocole d’extraction d’ARN :
Les embryons sont homogénéisés dans du Trizol à l’aide d’une seringue de 1 ml équipée d’une aiguille 21 G x 1/2. Du chloroforme saturé d’eau (200 µl) est ensuite ajouté aux échantillons et mélangé par vortex. Les échantillons sont centrifugés pendant 15 min à 13.000 g à 4 °C. La phase aqueuse au dessus est récupérée (les protéines restent dans la phase organique; l’ADN est en général à l’interface entre les deux phases) et on ajoute à 500 µl d’isopropanol pour précipiter l’ARN. L’ARN total est précipité par centrifugation à 13.000 x g pendant 30 min à 4 °C. Le culot est ensuite lavé avec de l’éthanol, centrifugé à nouveau et remis en suspension dans 50 µl d’eau exempte de RNAse. L’ADN résiduel dans les échantillons est éliminé en incubant les échantillons dans de la DNAse à 37 °C pendant 10 min. L’ARN total est purifié à partir de la réaction de DNAse en ajoutant 150 µl d’alcool isoamylique-phénol-chloroforme suivi d’une centrifugation à 13.000 x g pendant 5 min. La phase aqueuse supérieure est récupérée et mélangée avec 300 µl d’éthanol et 25 µl d’acétate d’ammonium (4 M pH 5,6) pour précipiter l’ARN. Enfin, l’ARN isolé est précipité par centrifugation pendant 15 min à 13.000 x g à 4 °C et les culots sont lavés dans de l’éthanol et remis en suspension dans 30 µl d’eau sans RNAse.
Ensuite, l’ARN est rétrotranscrit en ADN complémentaire (ADNc) grâce à une rétrotranscriptase. Puis des fragments spécifiques de cet ADNc sont amplifiés par PCR grâce à des couples d’amorces oligonucléotidiques. On choisit ces amorces dans la séquence codante des gènes d’intérêt.

Pour (re)voir le principe de la PCR : voir cette page
La PCR peut être classique, c’est-à-dire que l’on observe la quantité d’ADN obtenue après migration sur un gel d’électrophorèse ou la PCR peut être quantitative (qPCR ou aussi PCR en temps réel) signifiant que l’on observe la quantité d’ADN au cours de l’amplification grâce à une molécule qui devient fluorescente en présence d’ADN double brin (SYBRGreen).

La méthode de qPCR est quantitative car on vérifie que la quantité d’ADN a bien doublé à chaque cycle de PCR pour chaque couple d’oligonucléotides utilisé. Ce n’est pas le cas dans la PCR classique où l’on observe seulement le résultat final au bout de 30 à 40 cycles selon les protocoles.
Pour la RT-(q)PCR dans tous les cas, il faut inclure la mesure de l’expression d’un ou de plusieurs gènes de ménage (des gènes dont l’expression n’est pas censée bouger entre les différentes conditions pour servir de référence de normalisation).
RNAseq
L’hybridation in situ pour détecter les ARNm ou la RT-PCR sont des méthodes assez longue et laborieuses (il faut procéder gène par gène). Une méthode qui permettrait de détecter l’expression de plusieurs centaines ou de plusieurs milliers de gènes à la fois est la bienvenue.
Dans les années 2000, se sont développées les puces à ADN (microarray) mais les grands progrès du séquençage avec l’avènement du NGS (Next Generation Sequencing ou séquençage à haut débit) ont permis d’utiliser une méthode encore plus efficace (Goldman et Domschke, 2014). Le RNAseq tire parti des capacités de débit élevé de cette nouvelle méthode et permet aussi de quantifier les ARN présents dans un tissu. Plus précisément, les ARN sont extraits et purifiés à partir des échantillons et convertis en ADN complémentaire (ADNc) avec des procédures standard utilisant la transcriptase inverse. Jusqu’à présent, cela ressemble au début d’une RT-PCR. Mais pour le RNAseq, cet ADNc est fragmenté en morceaux plus petits et des séquences connues d’adaptateurs sont ajoutées aux extrémités. Ces adaptateurs permettent l’immobilisation et l’amplification par PCR de ces ADNc. Le séquençage de nouvelle génération peut analyser ces transcrits et les quantifier après alignement des séquences obtenues sur le génome (Goldman et Domschke 2014).
Le RNA-seq est particulièrement puissant pour comparer les transcriptomes entre des échantillons quasi-identiques ne différant que par certains paramètres expérimentaux (ajout on non d’un morphogène par exemple). Après une analyse statistique, on obtient une liste de sDEG pour significantly Differentially Expressed Genes. Le développement du tri cellulaire activé par la fluorescence (FACS) et de la microdissection a permis l’isolement précis des tissus et des cellules d’où sont extraits les ARN, compensant quelque peu l’absence de résolution spatiale de cette approche. Les progrès récents de la sensibilité du RNAseq ainsi que des méthodes de fluidique permettent désormais de réaliser une analyse transcriptomique de cellules uniques (single cell RNAseq ou scRNAseq) (Klein et al., 2015). Le terme « cellules isolées » serait sans doute plus approprié mais le terme « cellules uniques » est entré dans les mœurs.
Voir la vidéo d’explication sur ce site.


Source : https://www.nature.com/articles/s41467-021-23758-w
Cette méthode contourne les limites de l’expression génique « moyennée » à partir d’organes entiers ou d’échantillons de tissus et permet ainsi la définition et la caractérisation de cellules individuelles à haute résolution. Ce type d’étude a généralement permis de se rendre compte d’une diversité insoupçonnée de profils transcriptomiques au sein de tissus que l’on pensait homogènes et de connaitre de manière plus précise les étapes de transition au cours du temps d’une population cellulaire. Cette technique a cependant des limitations car seule la fraction la plus exprimée des transcrits peut être analysée, laissant de côté les gènes peu transcrits mais dont certains peuvent néanmoins avoir un impact significatif. Également, la résolution spatiale est perdue.
Ce genre d’analyse demande de solides bases en biostatistiques et en bioinformatique qui permettent de sortir les informations les plus pertinentes de la quantité énorme de données que peut générer ce genre d’étude. On peut classer par exemple les gènes différentiellement exprimés par fonction ou par localisation subcellulaire grâce à la nomenclature Gene Ontology (GO).
On peut aussi réaliser des clusters, c’est-à-dire des regroupements de gènes qui ont des profils d’expression similaires.

Des raffinements de l’analyse des résultats RNAseq permettent d’apporter une dynamique temporelle. Par exemple l’étude dite de vélocité d’ARN compare les ARN pré-messagers non épissés (les gènes les plus récemment transcrits) avec les ARN messagers épissés (les gènes transcrits il y a plus longtemps).

Signalons que jusqu’à présent les techniques transcriptomiques passaient toujours par une réverse-transcription et la production d’ADNc (ADN complémentaire) à partir des ARN. Il est désormais possible de séquencer directement les ARN par la technique du séquençage par nanopore (voir les détails avec ce lien).
Les gènes rapporteurs
Étudier l’expression d’un gène précis est parfois complexe alors que certains gènes produisent des protéines dont la présence et l’activité est aisément détectable. Ce sont les gènes rapporteurs. Les produits de ces gènes ne doivent pas être présent naturellement pour que le signal observé soit clair, et ils ne doivent pas interférer avec la physiologie cellulaire pour ne pas perturber le développement. Les gènes rapporteurs peuvent coder : soit des enzymes dont le produit est facilement détectable (lumière pour la luciférase, coloration bleu/rouge pour la β-galactosidase (selon le substrat Xgal/Redgal, respectivement), soit des protéines fluorescentes telles que la GFP et ses dérivés.


Une version commentée de cette figure est disponible en vidéo.
Source : https://www.cell.com/cell/fulltext/S0092-8674(00)80682-0
L’avantage des protéines fluorescentes est qu’elles permettent des observations sur les embryons vivants, illuminés avec la bonne longueur d’onde tandis que la coloration à la β-galactosidase nécessite généralement une fixation. Depuis récemment, l’activité luciférase peut aussi être observée sur des tissus vivants et non plus seulement dans des extraits protéiques.
Couplés avec de la transgénèse, les gènes rapporteurs peuvent être des outils puissants pour étudier les promoteurs et les enhancers puisque ces gènes peuvent être exprimés dans les embryons sous le contrôle de ces éléments. L’adéquation du patron d’expression du gène rapporteur avec l’expression du gène endogène dont on étudie les éléments de régulation transcriptionnelle permet de savoir si les fragments de promoteurs ou d’enhancers choisis sont pertinents ou non.
Exemple d’utilisation de gène rapporteur :

Les gènes rapporteurs peuvent aussi être introduits par knock-in dans le locus lui-même du gène et co-transcrit avec lui. Dans l’exemple qui suit, ce n’est pas tant l’étude des séquences régulatrices qui importe que de développer un outil pour repérer facilement l’expression d’un gène important qui est utile pour trier des cellules ou suivre un lignage particulier par exemple.

La fixation des facteurs de transcription à l’ADN
Hypersensitivité à la DNAse I
De manière assez indirecte, la fixation des facteurs de transcription peut se déduire de l’ouverture de la chromatine et donc de l’hypersensitivité de traitement à la DNAse I qui coupera plus souvent dans des régions chromatiniennes ouvertes.

ATACseq
Il s’agit d’une méthode qui tend à se substituer à l’hypersensibilité à la DNAse I. ATACseq vient de Assay for Transposase-Accessible Chromatin with highthroughput sequencing. Il s’agit de caractériser les régions accessibles de la chromatine en utilisant la propriété des transposons (éléments ADN mobiles) de s’intégrer préférentiellement dans les régions ouvertes de la chromatine. Le transposon recombinant Tn5 amène avec lui des séquences qui après fragmentation de l’ADN permettront de repérer les régions du génome où il s’est inséré.

Source : https://www.bioscience.co.uk/cpl/atac-seq-kit
Le protocole est plus rapide et demande moins de matériel que l’hypersensibilité à la DNAse I.
Des progrès techniques permettent maintenant de terminer l’accessibilité de la chromatine dans des cellules en provenance d’une coupe histologique ce qui permet de maintenir une information spatiale et de faire des cartes d’accessibilité de la chromatine pour un gène donné sur un embryon entier (en étudiant une série de coupes) (Deng et al., 2022).
EMSA (ElectroMobility Shift Assay) ou retard sur gel
Pour bien démontrer qu’un facteur de transcription (ou des histones ou tout autre protéine) interagit avec une séquence spécifique d’ADN, on peut utiliser le fait que la fixation de la protéine va retarder la migration de l’ADN dans une électrophorèse. Les protéines d’intérêt sont produites in vitro ou présentes dans des extraits nucléaires (on cherche des facteurs de transcription, il est logique de ne s’intéresser qu’à la fraction des protéines qui peut être au contact de l’ADN). Ces protéines sont mises à incuber avec des fragments d’ADN (d’une vingtaine à une centaines de pb) marqués (soit radioactivement mais cela ne se fait plus actuellement, soit avec de la biotine qui sera reconnue par la streptavidine couplée à l’enzyme HRP dont l’activité sera révélée par une réaction d’électroluminescence (ECL). On doit également mettre les mêmes fragments d’ADN mais non marqués pour réaliser une compétition et vérifier la spécificité de la liaison de la protéine d’intérêt à l’ADN ou on peut utiliser des fragments d’ADN mutés dans le site de fixation putatif que reconnaît la protéine comme témoin négatif (ou pour vérifier que ce site de fixation est bien effectif). On fait migrer l’ensemble sur un gel à électrophorèse mais en conditions non dénaturantes pour préserver la conformation des protéines et leurs interactions avec l’ADN. Ensuite, l’ensemble est transféré sur une membrane de nitrocellulose comme pour un western-blot puis le marqueur sur l’ADN est révélé. Voici un exemple ci-dessous :
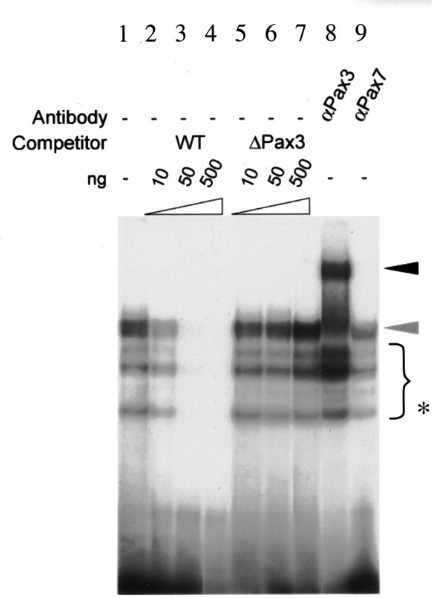
Les facteurs de transcription fixés à l’ADN ont été formellement identifiés grâce à l’utilisation d’anticorps dirigés contre eux car ils forment avec les facteurs de transcription et l’ADN un complexe encore plus gros qui ralentit d’autant plus la migration dans le gel lors de l’électrophorèse.
L’immunoprécipitation de la chromatine (ChIP)

D’après https://en.wikipedia.org/wiki/Chromatin_immunoprecipitation#/media/File:Chromatin_immunoprecipitation_sequencing.svg
Le CUT&Tag-seq
Une nouvelle technique vient se substituer de temps à autre au ChIPseq avec une sensibilité plus forte, moins de bruit de fond et moins de besoin en matériel de départ : le CUT&Tag-seq. Elle est toujours basée, comme la ChIPseq, sur la fixation d’un anticorps sur la protéine dont on veut savoir où elle se lie sur l’ADN génomique mais il n’y a plus d’immunoprécipitation. L’anticorps est reconnu par un complexe protéine A-transposase Tn5 et c’est la transposase qui coupe l’ADN à proximité de la séquence où la protéine d’intérêt s’est liée et y ajoute des tags (qui sont des adaptateurs pour le séquençage). Les fragments générés et qui sont taggés sont ensuite séquencés (Kaya-Okur et al., 2019).
Détection des modifications épigénétiques de la chromatine
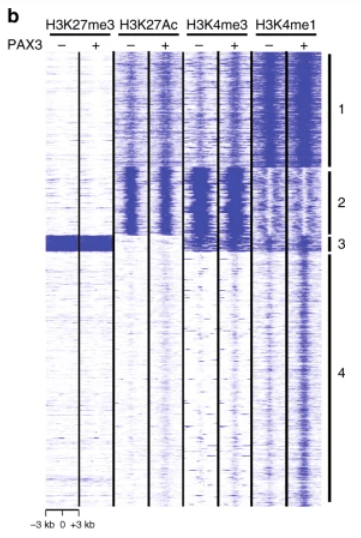
La méthode de ChIPseq précédemment décrite permet aussi de connaître où se trouvent dans le génome des formes méthylées ou acétylées d’histones particulières et donc d’avoir une idée de l’état favorable ou défavorable de la chromatine à la transcription. La reconnaissance par les anticorps est si spécifique que l’on peut distinguer une histone 3 diméthylée d’une histone 3 triméthylée.

Source : https://www.activemotif.com/catalog/details/91193/abflex-histone-h3k27ac-antibody-rab
La détection de la méthylation de l’ADN est expliquée sur ce lien. Citons le BS (Bisulfite sequencing) : L’ADN génomique est coupé par une ou plusieurs enzymes de restriction. On utilise fréquemment MspI qui n’est pas influencé par la méthylation de l’ADN. Les fragments sont ensuite traités avec le bisulfite. En présence de ce composé chimique, les cytosines non méthylées sont converties en uracile, alors que les cytosines méthylées ne sont pas affectées. L’ensemble des fragments traités est ensuite amplifié par PCR puis séquencé. On compare ensuite la séquence du fragment traité avec la séquence de l’ADN génomique : les cytosines qui étaient méthylées sont présentes sur les 2 séquences alors que celles qui n’étaient pas méthylées deviennent des uraciles (ou thymines après amplification PCR) dans le fragment traité au bisulfite.

Détection des structures chromatiniennes
Hi-C

Etude de la traduction
Si la régulation de la transcription a attiré l’essentiel des regards jusqu’à récemment, l’étude de la traduction devient de plus en plus importante, en relation avec la découverte de l’étendue des régulations par les microARN. Parmi les méthodes développées, citons le TRAP (Translating Ribosome Affinity Purification) qui permet de connaître quels ARNm sont effectivement traduits à un instant donné dans une population de cellules.

Pour l’étude de l’action des microARN sur la traduction, on peut avoir recours à la technique de séquestration de microARN. On utilise des « éponges à microARN » en faisant exprimer dans les cellules de multiples copies des séquences cibles d’un microARN donné pour qu’il s’associe à ces séquences et non pas aux ARNm qu’il cible habituellement (voir par exemple : https://www.accegen.com/services/microrna-sponge-service/).
Le résultat de la traduction : la présence de protéines (et aussi leur éventuelles modifications post-traductionnelles) peut s’étudier par western-blot ou immunfluorescence qui sont présentés sur cette page.
Modifications du génotype
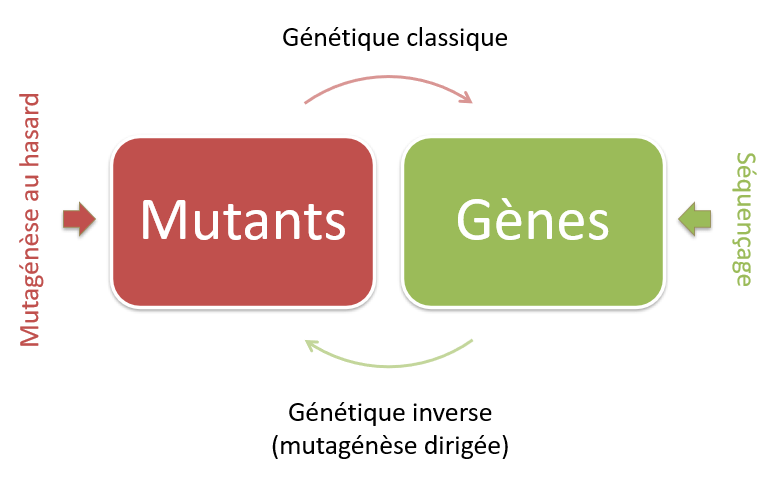
De manière historique, la génétique du développement a procédé par l’observation de mutants soit naturels, soit induits par des agents mutagènes et ensuite la cartographie génétique a permis de remonter aux loci impliqués puis aux séquences des gènes dès que la technique de séquençage Sanger a été mise au point en 1977. De célèbres criblages par mutagénèse chez la drosophile en 1980 (600 mutations obtenues réparties sur 120 gènes) et sur le poisson zèbre en 1996 ont permis l’identification de très nombreux gènes essentiels pour le développement.
Cependant, les techniques plus précises de génétique inverse se sont progressivement imposées, grâce à des procédures optimisées et de plus en plus précises de mutagénèse dirigée que nous allons voir. Elles consistent à introduire soit des mutations gain-de-fonction ou des perte-de-fonction dans des gènes déjà connus de par leur séquence. Les mutations gain-de-fonction permettent de savoir à quoi le produit du gène est suffisant et les mutation perte-de-fonction permettent de savoir à quoi le produit du gène est nécessaire.
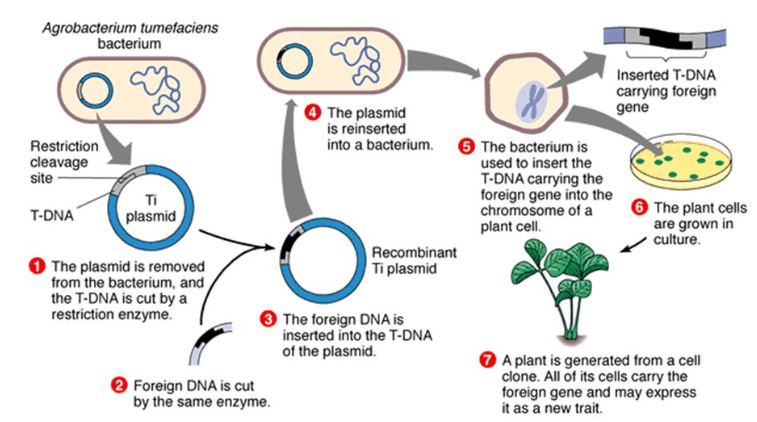
Transgénèse chez les plantes
Les propriétés de la bactérie pathogène Agrobacterium tumefaciens (qui provoque habituellement des galles) sont utilisées comme vecteur des gènes. On utilise le plus souvent des plasmides dérivés de son plasmide Ti dont une partie, l’ADN-T est habituellement transféré dans le génome de la plante. Pour la transgénèse, on utilise des plasmides Ti désarmés (ou D-Ti), car leur ADN ne porte plus les gènes responsables du pouvoir pathogène, et l’ADN-T contient les gènes que l’on souhaite étudier et un ou plusieurs gènes de sélection (le gène de résistance à la kanamycine par exemple). L’ADN-T modifié s’insère dans le génome et des agents de sélection permettent de ne faire survivre que les cellules végétales transformées. Des plantes entières sont ensuite régénérées à partir des protoplastes (cellules végétales sans parois) ou des cals où la transformation a eu lieu par des techniques classiques de culture in vitro, faisant intervenir les hormones végétales telles que l’auxine et les cytokinines.
La méthode « floral dip » permet de réaliser la transformation dans des bourgeons floraux en développement. Le transfert de l’ADN-T peut alors affecter des cellules germinales et le transgène sera transmis à la génération suivante par les gamétophytes (mâles ou femelles). On sélectionne alors quelles plantules issues de la fécondation et de la germination ont intégré le transgène.
Une autre méthode très différente pour créer des plantes transgéniques utilise la biolistique (mot fabriqué à partir de biologie et balistique) : des billes d’or ou de tungstène de 1 µm de diamètre sont enrobées d’ADN et projetées à grande vitesse vers des cellules végétales. Certaines traversent la paroi et la membrane plasmique et dans un petit nombre de cas l’ADN s’insère dans le génome (nucléaire mais aussi éventuellement mitochondrial ou chloroplastique).
Transgénèse chez la drosophile et système FRT-FLP
La transgénèse chez la drosophile consiste à insérer une séquence
d’ADN connue dans l’ADN chromosomique en utilisant comme vecteur un élément transposable (transposon) qui se transpose spontanément dans certaines souches de drosophiles. Ce transposon est connu sous le nom d’élément P. Les éléments P peuvent s’insérer dans n’importe quel site du génome et peuvent aussi se transposer d’un site à un autre dans les cellules germinales, une action qui réclame la présence d’une enzyme appelée transposase. Comme ce mécanisme est susceptible de générer de l’instabilité génomique, on a retiré aux éléments P servant de vecteur de transgénèse le gène codant la transposase. La transposase nécessaire à l’insertion initiale de l’élément P est fourni par un élément P dit « helper », qui ne peut pas s’insérer dans le génome, et est donc rapidement éliminé. Les éléments P « vecteur » et « helper » sont injectés ensemble dans la partie postérieure de l’œuf où se forment les cellules germinales. En plus du gène à insérer, l’élément P modifié a un gène marqueur tel que l’allèle sauvage du gène white. Dans ce cas, l’élément P est inséré chez des mouches homozygotes pour l’allèle mutant white- (qui ont des yeux blancs à la place des yeux rouges habituels de la drosophile sauvage). Les yeux rouges constituant un caractère dominant sur les yeux blancs, les mouches chez lesquelles l’élément P a été inséré et est exprimé, auront des yeux rouges et seront ainsi facilement repérées.
Un système basé sur la spécificité de l’activité des promoteurs et utilisant la transgénèse permet de ne faire exprimer un gène d’intérêt ou un gène rapporteur uniquement dans les cellules où un promoteur est actif. Il s’agit du système UAS-GAL4. GAL4 est un facteur de transcription activateur qui reconnait une séquence spécifique appelée UAS (pour Upstream Activation Sequence).

Chez la drosophile, des pertes et des gains-de-fonctions peuvent être obtenus grâce au système FLP-FRT. FLP (ou flippase) est une recombinase de levure qui est capable de recombiner deux séquences FRT (pour Flippase Recognition Target) et de déléter les séquences qui se trouvent entre deux séquences FRT.

Electroporation chez le poulet
L’électroporation chez l’embryon de poulet in ovo est une technique de transfection transitoire permettant d’introduire du matériel génétique étranger dans une partie des cellules de l’embryon et c’est particulièrement simple dans le faire dans le tube neural. Un champ électrique est appliqué sur les tissus embryonnaires, ce qui perturbe temporairement la membrane plasmique des cellules, créant des pores réversibles permettant l’entrée d’acides nucléiques dans le cytosol des cellules situés à proximité de l’électrode positive (anode) car les acides nucléiques sont chargés négativement. L’autre côté, proche de la cathode, sert de témoin car les acides nucléiques n’ont pas pénétré dans le cytosol des cellules.
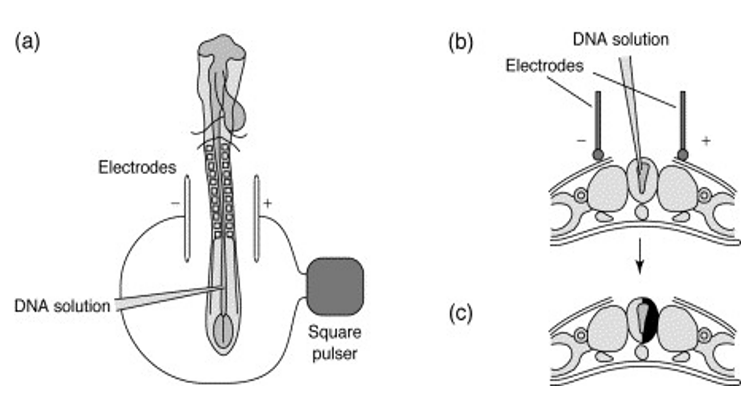
Les acides nucléiques électroporés peuvent être de l’ADN (un vecteur avec un gène sous le contrôle d’un promoteur spécifique par exemple) ou de l’ARN (par exemple un ARN interférent pour inhiber l’expression d’une protéine).
Transgénèse chez la souris
Par traitement hormonal, on fait superovuler des souris femelles puis on les accouple avec un mâle. Les zygotes sont rapidement récupérés dans les voies génitales puis incubés in vitro. L’ADN d’intérêt, souvent un gène sous le contrôle d’un promoteur spécifique est injecté dans le pronucléus mâle. Puis on sélectionne les embryons qui ont poursuivi correctement leur développement et on les injecte dans l’utérus d’une femelle pseudo-gestante (la copulation amène des stimuli mécaniques nécessaires au bon développement de l’utérus pour la gestation alors la femelle est préalablement accouplée avec mâle vasectomisé). Les souriceaux nés doivent ensuite être sélectionnés pour la présence et l’expression du transgène. En effet, l’insertion du transgène dans le génome ne réussit pas à chaque fois et le transgène peut aussi très bien s’être inséré dans de l’hétérochromatine silencieuse. On effectue une PCR, puis une RT-PCR ou alors un test qui permet de révéler l’expression d’un gène rapporteur si on en a mis un (coloration X-gal si on a mis le gène de la β-galactosidase). De toute manière, il faut toujours étudier plusieurs lignées transgéniques car l’ADN exogène s’insérant n’importe où au hasard dans le génome il faut vérifier que le phénotype observé n’est pas provoqué par la mutation introduite au point d’insertion mais bien par l’information du transgène lui-même.
Knock-out chez la souris
La technique du knock-out et du knock-in chez la souris est basé sur le remplacement d’un allèle sauvage par un allèle muté en utilisant la recombinaison homologue. Dans le cas du knock-out, le nouvel allèle ne donne pas de protéine fonctionnelle alors que dans le knock-in, une protéine au moins partiellement fonctionnelle (avec un site phosphorylable en moins par exemple) sera produite.

L’International Mouse Phenotypic Consortium ont maintenant généré des knock-out de plus de 6000 gènes chez la souris (Dickinson et al., 2016; Cacheiro et al., 2019)
Le système Cre-Lox
Les knock-out abolissent la fonctionnalité d’un gène depuis le début de son expression. Or parfois un gène peut avoir plusieurs fonctions successives à différents moments du développement. S’il a un rôle vital à une phase précoce du développement, un knock-out ne permettra pas de connaître sa fonction à des phases tardives. Le système Cre-Lox permet de franchir cet obstacle en rendant possible une délétion d’un gène contrôlée spatio-temporellement au cours du développement.
La recombinase Cre est une endonucléase du bactériophage P1 qui est capable d’exciser de l’ADN toutes séquences entre deux sites de quelques nucléotides appelés LoxP (plus précisément, les séquences LoxP sont constituées de 2 séquences inversées de 13 paires de bases séparées par 8 paires de bases).

Ainsi, on peut créer une souris transgénique exprimant la Cre sous certaines conditions :
- sous le contrôle d’un promoteur spécifique dont on connait l’activité spatio-temporelle
- activable par une injection de tamoxifène (analogue des œstrogènes) pour une forme de la Cre liée au domaine de fixation du ligand du récepteur aux œstrogènes. Pour le développement embryonnaire, le tamoxifène peut être injecté dans la mère et traverse le placenta pour activer la Cre dans l’embryon
- On peut combiner les deux approches précédentes.
On doit ensuite croiser la souris transgénique exprimant Cre de manière contrôlée avec une souris knock-in où les allèles endogènes ont été remplacés par des allèles flanqués de 2 séquences LoxP (on dit que l’allèle a été floxé). Le gène sera délété uniquement lorsque la Cre sera présente et fonctionnelle.
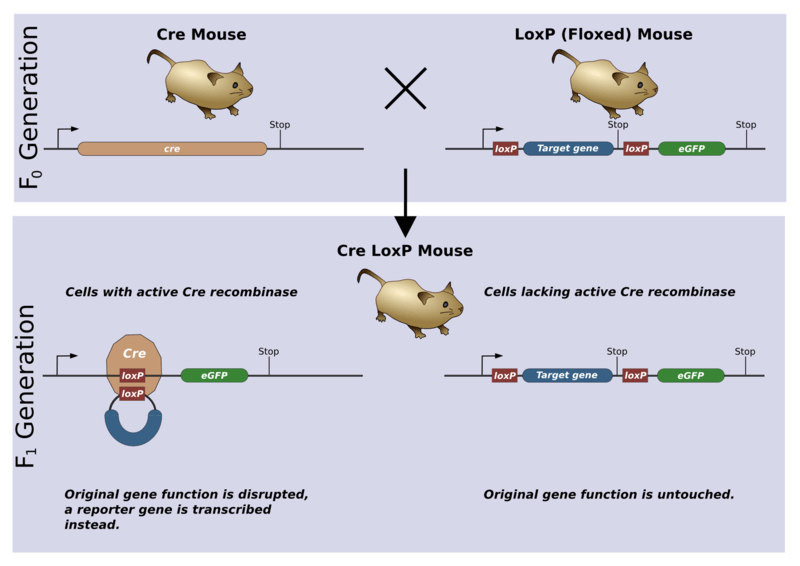
Le système Cre-Lox peut aussi être utilisé pour faire du suivi de lignage cellulaire. L’activation d’un promoteur donné permet l’expression de la Cre qui va réaliser la délétion d’une séquence générant un codon STOP qui empêche la production d’une protéine rapportrice fonctionnelle. Toutes les cellules qui ont activé le promoteur et aussi ses descendantes vont alors exprimer la protéine rapportrice. Cela est valable y compris pour les cellules où l’activité du promoteur se sera éteinte car la délétion de la séquence générant un codon STOP est définitive et donc une expression transitoire de la Cre suffit.
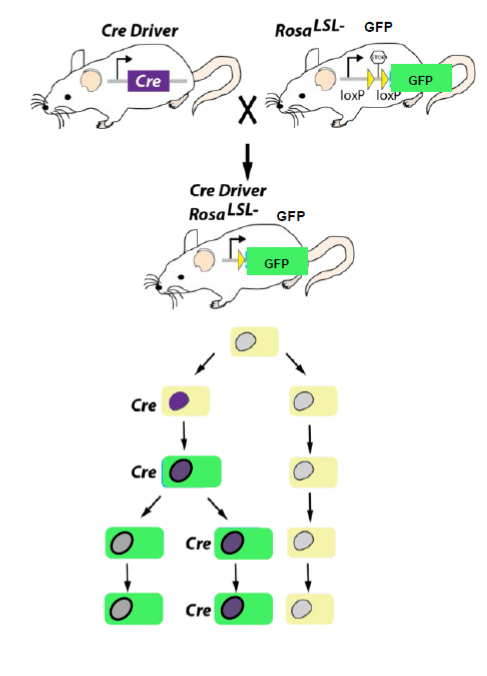
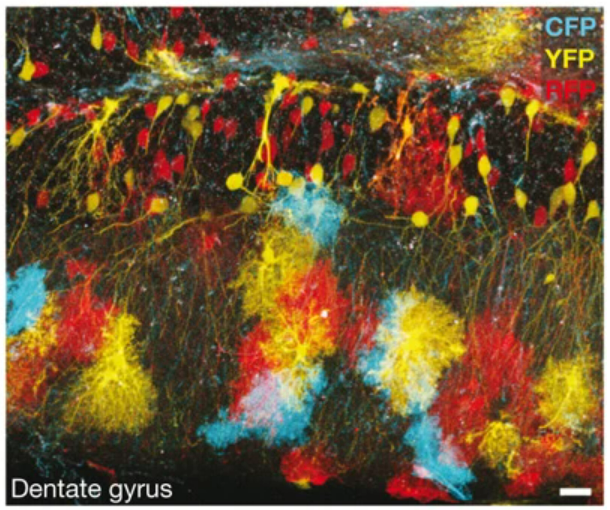
Une méthode basée sur le système Cre-Lox permet de suivre individuellement le devenir de plusieurs cellules en leur faisant exprimer une combinaison de fluorophores différents : la méthode Brainbow qui a été développée pour l’étude des connexions nerveuses dans le cerveau mais qui peut s’appliquer à d’autres systèmes.

CRISPR/Cas9
CRISPR-Cas9 a largement remplacé les méthodes traditionnelles de production d’animaux transgéniques et knock-out en raison de sa rapidité et de sa grande efficacité d’édition (Burgio, 2018). De manière plus transformative, CRISPR-Cas9 a élargi la portée des efforts pour comprendre comment les réseaux de gènes contrôlent l’acquisition du destin cellulaire.
Adapté d’un système « immunitaire » bactérien, le système CRISPR a été récupéré par les chercheurs pour une utilisation dans l’édition des génomes eucaryotes en fabriquant des ARN guides simples (sgRNA) qui se complexent avec une endonucléase, le plus souvent Cas9, pour induire des cassures double brin (DSB) à un emplacement spécifique dans le génome, spécifié par la séquence de sgRNA et un motif adjacent protospacer (PAM).
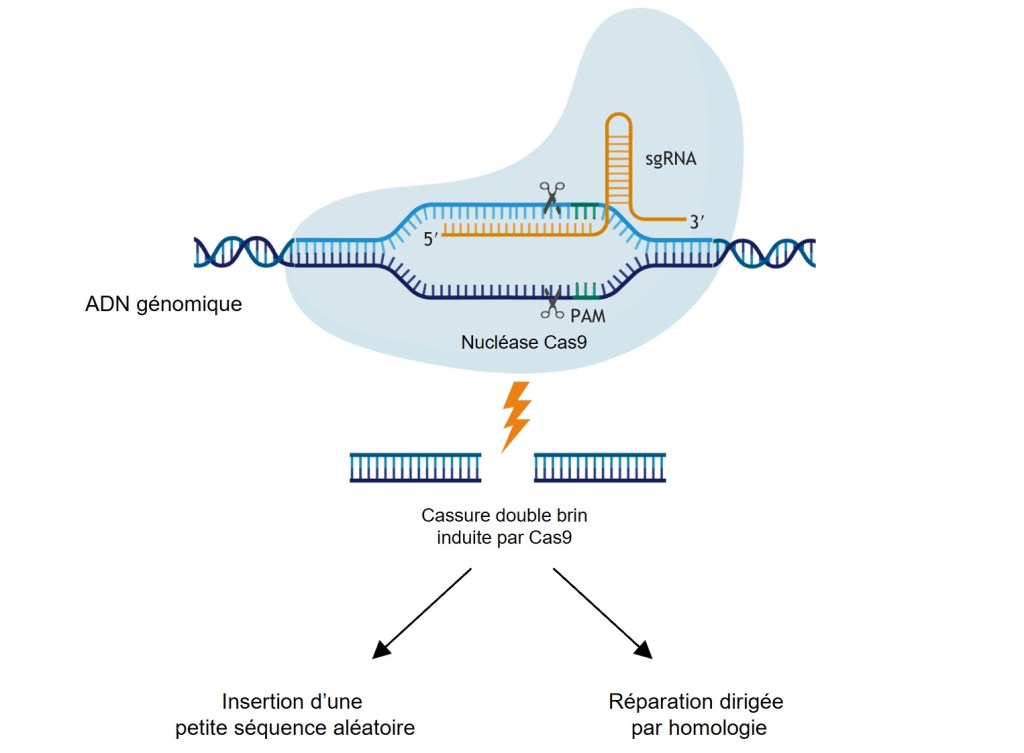
Après la cassure double brin, les enzymes de réparation de l’ADN de la cellule entrent en action. Elles peuvent combler la cassure :
- soit en insérant une petite séquence aléatoire (méthode de réparation appelée NHEJ pour Non-Homologous End Joining), ce qui introduit une mutation et par exemple un décalage du cadre de lecture si on se trouve dans la séquence codante d’un gène et que la séquence aléatoire insérée n’a pas un nombre de nucléotides multiple de 3.
- soit, en présence d’un fragment d’ADN exogène comportant des séquences homologues autour de la cassure et une séquence nouvelle que l’on souhaite insérer, remplacer par recombinaison homologue l’allèle abîmé par la cassure par l’allèle que l’on a introduit. Le processus s’appelle HDR pour Homologous Directed Repair. Il permet de faire une mutagénèse dirigée très précise et permet d’introduire par exemple une mutation ponctuelle sur un site de phosphorylation (qui changera une sérine ou une tyrosine en alanine), sur un site de fixation d’un facteur de transcription sur un promoteur, ou carrément l’introduction d’un gène rapporteur sur le site de la cassure double brin.

Initialement, l’édition CRISPR était limitée aux séquences proximales du PAM (protospacer adjacent motif, une courte séquence d’ADN (généralement de 2 à 6 paires de bases) qui suit la région d’ADN ciblée pour le clivage par le CRISPR). Cependant, désormais, de nombreux variants Cas9 et Cas12a de bactéries et de phages ont été caractérisés, chacun avec des PAM distincts, ce qui permet d’élargir les cibles possibles à des régions plus variées avec des PAM différents (Manghwar et al., 2019; Pausch et al., 2020). En outre, les variantes Cas9 ont été conçues avec une reconnaissance PAM «plus lâche» qui sert à encore élargir la plage de ciblage (Kleinstiver et al., 2015; Nishimasu et al., 2018; Walton et al., 2020).
Le principal danger de la méthode CRISPR/Cas9 est constitué par la formation de mutations ailleurs dans le génome qu’à l’endroit prévu. Des variantes avec une réduction de l’édition de l’ADN hors cible ont été développées, bien qu’elles réduisent simultanément l’efficacité de l’édition (Kim et al., 2020; Kleinstiver et al., 2016; Slaymaker et al., 2016).
Les facteurs épigénétiques, tels que les modifications post-transcriptionnelles des histones, la méthylation de l’ADN et les ARN non codants, contribuent énormément au contrôle de l’expression génétique. Disséquer le rôle de ces modifications épigénétiques sur la régulation des gènes a été accéléré par la capacité à modifier spécifiquement les modifications épigénétiques en utilisant CRISPR/dCas9. dCas9 est un variant de Cas9 qui joue juste le rôle de guide mais qui ne clive pas l’ADN car son activité endonucléase est abolie par des mutations (D10A et H840A). Ce variant a été utilisé pour recruter des modificateurs épigénétiques afin d’évaluer leurs conséquences fonctionnelles. dCas9 est alors fusionné à des enzymes et des complexes de modification épigénétique, par exemple l’histone déméthylase 1A spécifique de la lysine (LSD1; KDM1A) (Kearns et al., 2015), l’histone acétyltransférase p300 (Hilton et al ., 2015), ou EZH2, un répresseur de la famille Polycomb (O’Geen et al., 2019).

b) Expression relative de l’ARNm d’IL1RN (Interleukin 1 Receptor Antagonist), de MYOD et d’OCT4, déterminée par qRT-PCR, avec chaque région promotrice respective ciblée par les diverses protéines fusions
co-transfectée avec quatre ARNg. Les nombres au-dessus des barres indiquent l’expression moyenne ; NLS = séquence de localisation nucléaire; HA = étiquette d’épitope d’hémagglutinine (pour l’étude de l’expression des protéines); CH = région riche en cystéine histidine; Bd = bromodomaine; HAT = domaine histone acétyltransférase. On constate une nette augmentation de l’expression des gènes avec la forme p300Core en relation avec son activité HAT (quelque chose de pas encore bien compris inhibe l’activité HAT avec la protéine entière). Source : https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4430400/pdf/nihms672873.pdf
dCas9 fusionnée à des activateurs de la transcription (comme VP64) a été aussi utilisé pour produire des cellules iPS et pour la transdifférenciation de fibroblastes en neurones (Black et al., 2016; Liu et al., 2018; Weltner et al., 2018).

L’efficacité de la production des iPS a été augmentée en activant non seulement les facteurs de transcription connus (POU5F1, MYC, KLF4, SOX2 et LIN28A), mais aussi en se fixant sur des séquences Alu enrichis en motif EGA répétitif (motif EEA), qui se trouvent souvent sur les promoteurs de gènes exprimés lors de l’activation de la transcription du génome de l’embryon humain, révélant ainsi un avantage supplémentaire de dCas9 qui ne peut pas être facilement réalisé à l’aide de transgènes. Néanmoins, dans tous ces paradigmes, la reprogrammation basée sur dCas9 est restée nettement moins puissante que la reprogrammation basée sur les transgènes, probablement parce que les niveaux d’induction de facteurs de transcription requis pour une reprogrammation efficace ne peuvent pas être atteints avec la technologie CRISPR actuelle.
En ce qui concerne l’utilisation de la technique de CRISPR/Cas9 en thérapie génique, il faut s’armer de prudence concernant le ciblage de l’édition qui n’est parfois pas correct. Une étude a montré que des éditions dans les cellules des embryons humains précoces avaient provoqué des mutations non souhaitées dans 22% des cellules concernées. Bien qu’il y ait eu des améliorations, une instabilité génomique préoccupante peut suivre un traitement CRISPR/Cas9 (Papathanasiou et al., 2021).
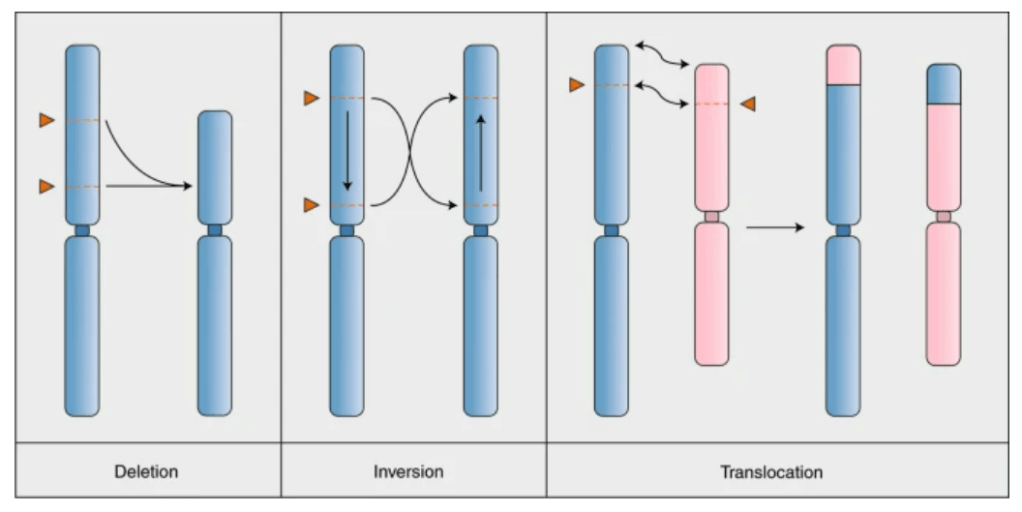
En dehors des mutations « ponctuelles » ou limitées que nous venons de voir, CRISPR/Cas9 peut aussi être utilisé pour des modifications génomiques à plus grande échelle. Si deux cassures double brin sont induites simultanément sur le même chromosome ou sur des chromosomes différents, des réarrangements chromosomiques, tels que des inversions et des translocations, peuvent être induits. Chez les plantes, les réarrangements chromosomiques contribuent fortement aux processus évolutifs et sont répandus dans diverses familles de plantes et les généticiens des plantes voient un grand intérêt dans CRISPR/Cas9 également pour cette raison.
Signalons qu’il existe aussi une forme photoactivable de Cas9 qui permet de déclencher le changement d’allèle à un moment bien précis par une illumination LED (Takao et al., 2020).
Focus sur la réparation de l’ADN après la cassure double brin introduite par CRISPR/Cas9 :

Interférence ARN
Les principes de l’interférence ont été découverts par hasard au cours de l’étude de la coloration des pétunias au début des années 1990 puis a été développé chez le nématode Caenorhabditis elegans. De petits fragments d’ARN de 21-25 nucléotides appelés ARNi se lient à une séquence spécifique complémentaire sur un ARNm et bloquent leur traduction et même leur transcription (un phénomène appelé RITS pour RNA-induced transcription silencing). Ces mécanismes se sont développés au cours de l’évolution pour s’opposer aux virus et aux transposons, notamment chez les plantes. Les chercheurs peuvent désormais les détourner à leur profit, non seulement pour inhiber la production d’une protéine dans les plantes mais aussi dans les cellules animales car les ARNi « imitent » chez eux des inhibiteurs de la traduction appelés microARN.
Chez les végétaux, on peut faire produire des ARNi par la plante grâce à la technique VIGS pour Virus-Induced Gene Silencing : un vecteur viral (généralement dérivé du virus TRV (Tobacco Rattle Virus)) est introduit dans la plante et permet de produire des ARN double brin contenant une partie de la séquence de l’ARNm dont il faut inhiber la traduction. Les cellules végétales produisent ensuite elles-mêmes les ARNi nécessaires dans un mécanisme de défense, croyant que cette séquence est une séquence virale alors qu’elle correspond en fait à l’un de ses propres ARNm. C’est une méthode plus efficace et rapide que d’introduire des ARNi dans les cellules végétales.
Pour introduire des ARNi dans C. elegans, il suffit de leur donner à manger des bactéries E. coli qui expriment les ARNi sous la forme d’un double brin (plus stable). Par un mécanisme peu compris, ces ARN double brin sont capables d’atteindre toutes les cellules où la machinerie cellulaire se charge de les transformer en ARNi simple brin. L’introduction des ARNi n’est pas aussi simple dans les autres systèmes. Dans les cellules animales en culture, on peut les introduire par lipofection. Dans les embryons, on peut les introduire par électroporation. Mais leur présence est alors transitoire. Pour une présence plus prolongée on peut introduire dans les cellules un ADN qui code un ARN qui va donner un ARNi. Cet ARN est appelé shARN (sh pour short hairpin) car on lui donne une séquence qui le fait prendre une structure secondaire repliée pour être plus stable et mieux se faire reconnaître par la protéine Dicer qui va le confondre avec un précurseur de microARN et va le transformer en ARNi.
Des criblages peuvent être réalisés grâce à des banques de ARNi ou de shARN. Cela permet, de manière non biaisée, de trouver des nouveaux candidats pour une fonction donnée comme dans l’exemple suivant :

Chez le poisson-zèbre et le xénope, on peut utiliser une technique similaire à l’ARN interférent. L’expression des gènes dans les embryons de xénope peut être facilement manipulée à l’aide d’oligonucléotides morpholino antisens (MO) pour une perte de fonction. Les MO sont des polymères avec une séquence de bases azotées mais ces bases ne sont pas accrochées à des (désoxy)riboses mais à des morpholines qui sont liées entre eux par des groupements phosphorodiamidate au lieu de groupements phosphate. Cette structure est particulièrement résistante à la dégradation. Les MO sont injectés dans les cellules au cours du développement précoce (quelquefois même chez le zygote). Les MO sont conçus pour cibler soit le site de départ de la traduction (le codon d’intiation AUG), soit un site d’épissage du transcrit primaire de l’ARNm cible.


Les outils bioinformatiques
L’accumulation exponentielle de données en provenance du séquençage et des analyses transcriptomiques a amené à développer de nombreux outils pour trouver des informations pertinentes. Si certains de ses outils sont une affaire de spécialistes, certains peuvent être utilisés de manière courante avec un minimum de formation :
BLAST : pour comparer des séquences entre elles ou une séquence avec les génomes sequencés.
Ensembl : géré par le European Bioinformatics Institute, il répertorie les principaux génomes d’organismes-modèles. Le serveur offre la possibilité de naviguer depuis le niveau du caryotype jusqu’au niveau de la séquence nucléique et de sa traduction dans les différentes phases de lecture.
geWorkbench : plateforme open-source avec de nombreux outils et de plug-ins pour des analyses génomiques et transcriptomiques.
MirGeneDB : une base de données des microARN chez de nombreuses espèces.
Reactome : base de données sur les voies de signalisation et leurs liens avec des processus cellulaires.
RCSB Protein Data Bank : site répertoriant les structures 3D des macromolécules (protéines mais aussi acides nucléiques, complexes nucléoprotéiques, sucres…)
AUTRES RESSOURCES SUR LES TECHNIQUES DE GENETIQUE
Présentation de nombreuses techniques avec pour cadre le développement du cerveau de la souris (RNBio – Sciences Sorbonne Université). Cliquez après la vidéo sur sommaire.
LIEN VERS LE GLOSSAIRE DES TERMES LIES A LA GENETIQUE
- Adhérences cellule-cellule
- Arabidopsis thaliana
- Axe antéro-postérieur chez la drosophile
- Biomécanique du développement
- Caenorhabditis elegans
- Concepts principaux
- Contrôle de la traduction
- Contrôle de la transcription
- Contrôle génétique et épigénétique
- Croissance du tube pollinique et double fécondation chez les Angiospermes
- Croissance et guidage axonal
- Des modèles animaux moins classiques
- Développement de l’oeil des Vertébrés
- Développement et évolution
- Et l’Humain ?
- Exercices sur l’ovogenèse, la spermatogenèse et la fécondation
- Exercices sur le contrôle de l’expression des gènes
- Exercices sur le développement des bourgeons de membre
- Exercices sur le développement des muscles striés squelettiques
- Exercices sur le développement des végétaux et les hormones végétales
- Exercices sur les cycles et les divisions cellulaires
- Exercices sur les étapes du développement, les inductions embryonnaires et la mise en place des axes de polarité
- Exercices sur les matrices extracellulaires, le cytosquelette et les adhérences cellule-cellule
- Exercices sur les voies de signalisation
- Glossaire
- Glossaire des termes liés à la génétique
- Glossaire des termes liés au cytosquelette, la matrice extracellulaire, l’adhérence et la migration cellulaire
- Hématopoïèse et développement des cellules du système immunitaire
- Histoire de la biologie cellulaire et de la biologie du développement
- L’acide rétinoïque
- L’apoptose
- L’autophagie
- L’organogenèse
- L’ovogénèse prépare le développement embryonnaire
- La drosophile
- La fécondation
- La formation des somites
- La gastrulation
- La gastrulation (version allégée)
- La métamorphose chez les Hexapodes et les Amphibiens
- La neurogénèse chez les mammifères adultes
- La neurulation
- La poule
- La signalisation calcique
- La souris
- La superfamille TGFβ et ses voies de signalisation
- La voie de signalisation de l’auxine et ses rôles
- La voie de signalisation Hedgehog
- La voie de signalisation Hippo et ses composants YAP/TAZ
- La voie de signalisation Notch
- Le clivage
- Le cytosquelette
- Le destin des cellules et les réseaux de régulation génique
- Le développement des bourgeons de membre
- Le développement des muscles striés squelettiques
- Le développement des organes génitaux et des cellules germinales
- Le développement du cortex
- Le méristème apical caulinaire en phase végétative et lors de la formation d’une fleur
- Le poisson zèbre
- Le xénope
- Les cellules des crêtes neurales
- Les cellules et les gènes en action dans le développement
- Les cellules souches
- Les cellules tumorales
- Les cycles et les divisions cellulaires
- Les étapes du développement
- Les étapes du développement embryonnaire d’Arabidopsis thaliana et leur contrôle
- Les inductions embryonnaires et les gradients de morphogène
- Les matrices extracellulaires animales
- Les organismes modèles
- Les parois des cellules végétales
- Les techniques et les outils pour la biologie cellulaire
- Les transitions épithélio-mésenchymateuses et les migrations cellulaires
- Les vésicules extracellulaires
- Les voies de signalisation
- Les voies de signalisation FGF
- Mise en place des axes chez les Vertébrés
- Structures et processus cellulaires
- Voies de signalisation WNT