Par Patrick Pla, Université Paris-Saclay
Au cours du développement des organismes pluricellulaires, les cellules ont des destins différents, générant une diversité de cellules avec une structure et des fonctions bien précises. On considère qu’il existe plus de 800 types cellulaires dans un corps humain (Han et al., 2020). Générer une telle diversité qui aboutit à un organisme fonctionnel nécessite un contrôle étroit.
Le zygote donne, par définition, naissance à l’ensemble des cellules d’un embryon (et même dans certains cas à ses annexes embryonnaires). Il est considéré comme totipotent. Au fur et à mesure que les étapes du développement se déroulent, les cellules sont progressivement déterminées : même isolées de l’embryon ou déplacées dans l’embryon, elles finissent par donner les mêmes cellules différenciées que si on les avait laissées à leur place. Un programme génétique menant à la différenciation a été sélectionné de manière irréversible.
Les cellules embryonnaires passent donc par une étape pluripotente (capables de donner tous les types cellulaires appartenant aux 3 feuillets sauf quelques types) puis multipotente (capables de donner un grand nombre de types cellulaires restreints à un ou deux feuillets) avant de voir leurs potentialités se restreindre encore plus. Une représentation visuelle de ce processus est apportée par le paysage de Waddington, dans lequel la trajectoire de différenciation d’une cellule est conçue comme une boule roulant dans un paysage de vallées en bifurcation (Waddington, 1957). Dans cette représentation, le paysage est façonné par les réseaux de régulation génétique avec les vallées représentant les destins cellulaires. Les signaux de développement attribuent l’identité cellulaire en déterminant la vallée dans laquelle une cellule pénètre.

Comment cette restriction progressive du destin des cellules est-elle contrôlée ?

Comme le démontre l’expérience de clonage de John Gurdon réalisée en 1962 (Prix Nobel en 2012), le développement embryonnaire et la différenciation cellulaire n’entraînent pas de pertes quantitatives d’information génétique. En effet, l’ADN d’une cellule différenciée donnée peut diriger le développement de centaines de types cellulaires différents. Cela ne se fait pas sans difficultés : la plupart des clones ne se développent pas complètement (moins de 2% sont arrivés à l’âge adulte) mais l’important est que cela est possible.
Des équivalents de cette expérience de clonage ont été réalisés chez les Mammifères, d’abord chez la brebis en 1996 (brebis Dolly, seule survivante parmi les 434 essais de clonage réalisé par l’écossais Ian Wilmut) puis chez la souris en 1997. En 2003, une mule qui est un animal stérile a été cloné (Woods et al., 2003).
Même le noyau d’une cellule différenciée, remis dans un contexte adéquat (ici l’ovocyte) peut donc se révéler être totipotent. Au cours du développement embryonnaire, tout dépend donc de la manière dont les gènes sont exprimés. Le destin des cellules dépend ainsi de la cascade de régulations géniques qui démarre quelquefois avant même la fécondation (au cours de l’ovogenèse pour les gènes dits à effet maternel) et qui se poursuit tout au long du développement.
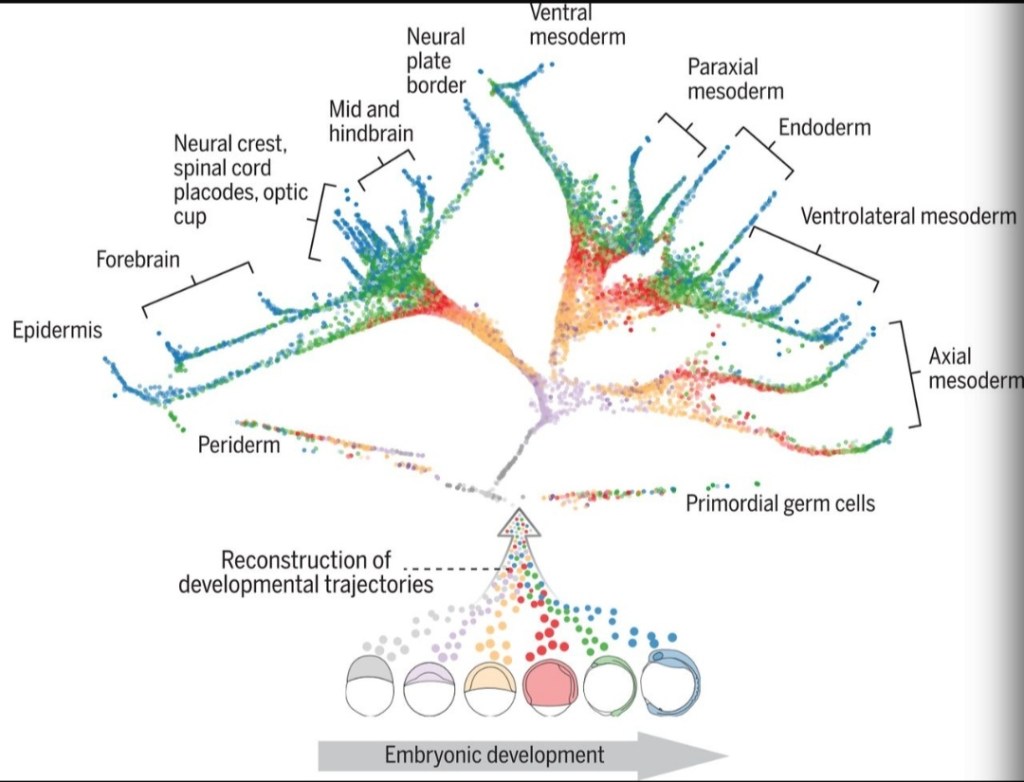

Les embryons sont caractérisés par des schémas de différenciation cellulaire très organisés et reproductibles. Par exemple, on peut observer les limites nettes de l’expression des gènes dans de nombreux tissus en développement. Ces limites définissent des territoires bien précis qui se développent souvent en des structures différentes. Les devenirs cellulaires sont déterminés par des réseaux de régulation de gènes (RRG), coordonnés dans l’espace par des signaux sécrétés (Davidson, 2010).
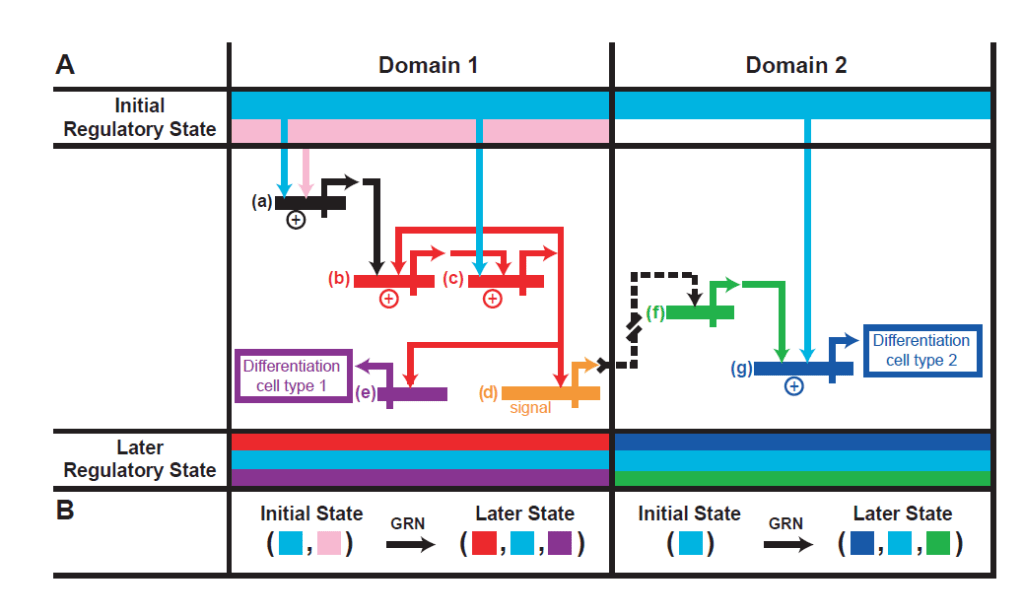

Il s’agit de comprendre comment la précision des destins cellulaires est obtenue malgré le « bruit de fond » biologique et les fluctuations stochastiques de la régulation de l’expression des gènes (Raser et O’Shea, 2005). Ces fluctuations peuvent d’ailleurs être nécessaires pour la mise en place de la répartition de certains types cellulaires au sein d’un tissu (Meyer et al., 2017).
Une vision moderne consiste à utiliser la théorie des systèmes dynamiques et à considérer les vallées de Waddington comme des états attracteurs du RRG (Enver et al., 2009; Balázsi et al., 2011). De ce point de vue, les cellules ne sont pas déterminées de manière linéaire dans le temps mais peuvent osciller temporairement entre un ou plusieurs états. Elles peuvent être chassées d’une vallée vers un attracteur adjacent, produisant ainsi un changement d’identité, non seulement par des signaux de développement mais également par les fluctuations naturelles de l’expression génétique. Ces fluctuations sont ensuite stabilisées, ce qui scelle le destin des cellules.
Comment les fluctuations sont-elles tamponnées dans les populations de cellules en développement pour garantir que les signaux de développement génèrent des patrons d’expression génique précis et reproductibles ? Lors de la régulation de la transcription, l’activité des éléments régulateurs redondants, l’architecture tridimensionnelle du génome, la présence d’allèles multiples amortissent les fluctuations et augmentent la robustesse de l’expression des gènes (Frankel et al., 2010; Lagha et al., 2012; Little et al., 2013; Tsai et al., 2019).

Au niveau du tissu, les mécanismes de régulation de la forme, de la pente ou de la variation des gradients des morphogènes ont été explorés et leurs effets sur la précision de l’expression des gènes détaillés (Sokolowski et al., 2012; Zagorski et al., 2017). Plusieurs mécanismes, parmi lesquels l’adhérence différentielle, les barrières mécaniques et la signalisation juxtacrine corrigent les anomalies et améliorent la précision, une fois la structuration tissulaire initiée (Dahmann et al., 2011; Addison et al., 2018).
VOIR LE CHAPITRE SUR LES MORPHOGENES ET LES INDUCTIONS EMBRYONNAIRES
Dans les premières étapes de l’auto-organisation, il est nécessaire d’établir une asymétrie et, par conséquent, de rompre la symétrie initiale de la population de cellules. La rupture de symétrie peut être induite par des fluctuations aléatoires de l’expression des gènes ou par des facteurs externes eux-mêmes asymétriques qui induisent une population hétérogène. Chez le xénope, la rupture de la symétrie est provoquée par la localisation de l’entrée du spermatozoïde. Le centriole qu’il apporte réorganise le réseau de microtubules ce qui provoque la rotation corticale. En conséquence, des déterminants dits « dorsaux » (ARNm et protéines) se déplacent d’un côté de l’embryon et deviennent absents de l’autre. La cascade de régulation génique déclenchée par ces déterminants sera différente de celle déclenchée de l’autre côté, le futur côté ventral.
Dans les embryons de mammifères, la symétrie est rompue lors de la ségrégation des premiers lignages, au cours de laquelle le trophectoderme et la masse cellulaire interne (MCI) sont établis. La signalisation Yap1 est importante dans ce contexte : elle est active dans les cellules externes de l’embryon, en initiant la transcription de Cdx2 qui est nécessaire à la formation du trophectoderme (Anani et al., 2014 ; Maître et al., 2016).
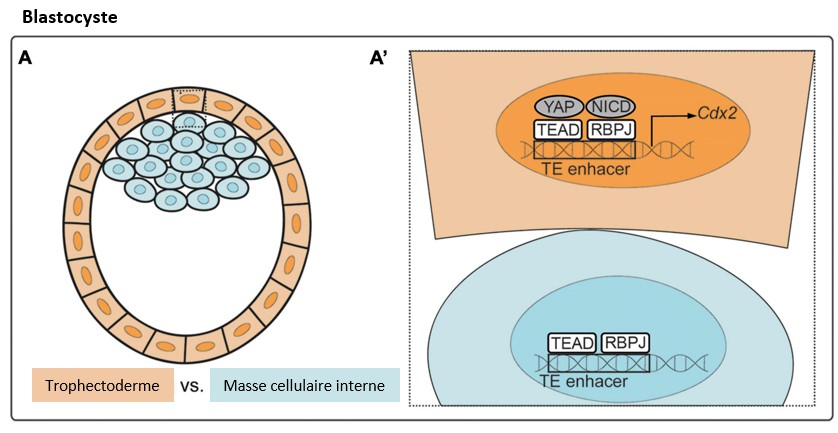
Une version commentée de cette figure est disponible sur cette vidéo.
Source : https://www.frontiersin.org/articles/10.3389/fcell.2021.711531/full

La voie Hippo, qui comprend Yap1, joue un rôle important dans l’adhésion et la polarité cellulaires et est activée par mécanotransduction. La position périphérique des cellules du trophectoderme provoque sans doute des tensions mécaniques à l’origine de l’activation de Yap1.
Suivant la spécification de la masse cellulaire interne (MCI), une autre subdivision se produit en son sein : en épiblaste et endoderme primitif.
Etudions comment se passe la répartition entre ces deux destinées dans la MCI. A E3,25 jours de développement, toutes les cellules de la MCI expriment uniformément les facteurs de transcriptions épiblastiques impliqués dans le maintien de la pluripotence Sox2, Oct4 et Nanog, ainsi que le facteur de transcription qui sera crucial pour le développement de l’endoderme primitif GATA6 (Dietrich et Hiiragi, 2007 ; Plusa et al., 2008). A E3.5 jours de développement, la MCI perd son uniformité et commence à présenter une distribution poivre-et-sel de cellules exprimant soit Nanog soit GATA6. A E4.5 jours, les deux lignées sont spécifiées. Les cellules de l’endoderme primitif se séparent pour former une couche épithéliale qui entoure l’épiblaste pluripotent. Les cellules exprimant GATA6, et ensuite ses cibles Sox17 et GATA4, activent un réseau transcriptionnel favorisant l’engagement dans le lignage de l’endoderme primitif (Artus et al., 2011 ; Bessonnard et al., 2014 ; Koutsourakis et al., 1999 ; Morrisey et al., 1998; Schrode et al., 2014). Cependant, ces précurseurs peuvent encore changer et devenir des cellules épiblastiques si le ratio des cellules épiblaste/endoderme primitif est affecté (Saiz et al., 2020; Yamanaka et al., 2010), indiquant que les cellules à ce stade ont un encore un comportement plastique et pas complètement engagé. Cette adaptabilité est perdue dans le blastocyste tardif (E5) suggérant que les destins de l’endoderme primitif et de l’épiblaste sont irréversiblement engagés à ce stade (Grabarek et al., 2012; Yamanaka et al., 2010).
Pour caractériser l’état plastique des cellules de la MCI bipotentes, des cellules ES portant un transgène GATA6 inductible (les cellules ES n’expriment pas naturellement GATA6) ont été caractérisées. Dans un premier temps, GATA6 ne réprime pas l’expression de Nanog. Au cours de cette étape de coexpression, GATA6 lie des éléments de régulation (CRE) dans les promoteurs et les enhancers des gènes typiques de l’endoderme primitif et fonctionne comme un facteur de transcription pionnier pour les rendre accessibles pour une activation transcriptionnelle plus forte ultérieurement. Simultanément, GATA6 se lie à une fraction étonnamment grande des promoteurs contrôlant les gènes typiquement épiblastiques, conduisant à une co-occupation avec les facteurs de transcription de pluripotence, Nanog et Sox2. Dans un deuxième temps, si on oriente les cellules vers un destin d’endoderme primitif, GATA6 provoque l’éviction de Nanog et Sox2. Cet exemple montre comment la détermination des cellules se fait progressivement avec les mécanismes moléculaires associés.
Qu’est-ce qui provoque la relative irréversibilité d’une différenciation ? Les analyses épigénomiques de la différenciation dans les différents lignages à partir des cellules pluripotentes (ES ou iPS) mettent en évidence un programme dans lequel des facteurs de transcription spécifiques à la lignée sont induits puis renforcés avec en parallèle une répression de l’expression des gènes de pluripotence (Gifford et al., 2013, Suelves et al., 2016).
La structuration des réseaux de régulation géniques permet une progression dans la détermination vers la différenciation avec différentes modalités.
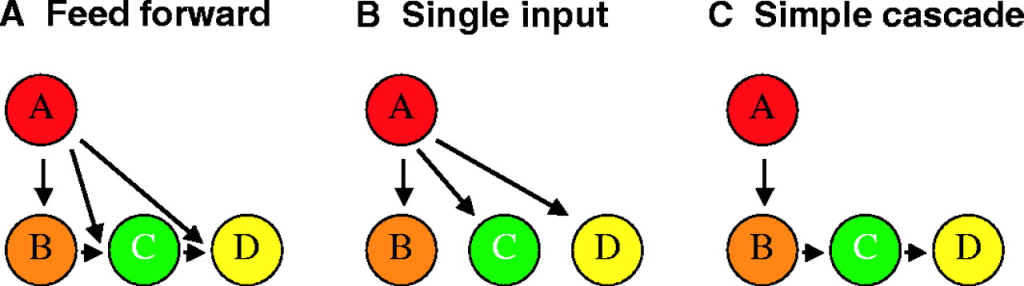
La détermination des destins cellulaires dépend d’une intégration de voies de signalisation pour activer l’expression de gènes clés. Par exemple, lors de la détermination de cellules pluripotentes iPS ou ES en précurseus méso-endodermiques, il faut d’abord faire agir la voie Wnt (processus de « priming ») et ensuite la voie activine/Nodal pour obtenir le rendement le plus élevé d’activation de l’expression du gène EOMES (Yoney et al., 2022).

Les mécanismes épigénétiques associés à la répression de la pluripotence ou des programmes génétiques alternatifs à un lignage dans lequel s’engage définitivement une cellule comprennent la méthylation de l’ADN et des méthylations répressives des histones telles que H3K27me3 et H3K9me3 (Feldman et al., 2006; Nicetto et Zaret, 2019). Ces modifications offrent un moyen de faire taire de façon permanente la transcription des gènes exprimés en cas de pluripotence ou dans un autre lignage, y compris en l’absence des signaux inducteurs initiaux de la détermination et de la différenciation. Les marques épigénétiques permettent ainsi d’établir une mémoire du destin cellulaire.

Le remodelage de la chromatine accompagnant les modifications épigénétiques est une étape essentielle qui est souvent réalisée par des protéines de type SWI/SNF. La composition des complexes contenant ces protéines peut varier au cours de la différenciation. Par exemple, au cours de la neurogenèse, les progéniteurs neuronaux possèdent le complexe de type SWI/SNF npBAF, qui comprend SS18, DPF2 et ACTL6A (BAF53a). Lors de la différenciation finale en neurones, plusieurs sous-unités sont échangées pour devenir le complexe neuronal BAF (nBAF), qui est plutôt composé de SS18L1 (CREST), DPF1/3 et ACTL6B (BAF53b) (Lessard et al., 2007).
Pour voir en détail les mécanismes génétiques et épigénétiques du contrôle de la transcription, voir cette page.
Les cas de maintien de la pluripotence
La pluripotence est un état dans lequel les cellules peuvent s’auto-renouveler et rester indifférenciées, conservant la capacité de donner naissance à des dérivés de n’importe quel feuillet embryonnaire. Cet état cellulaire est maintenu non seulement par un défaut d’expression des facteurs de détermination et de différenciation, mais aussi par un réseau de régulation génétique (RRG) complexe qui est étroitement contrôlé par un ensemble de facteurs de transcription : Nanog, Oct4 et Sox2 (Navarro et al., 2012; Rodda et al., 2005; Teo et al., 2011; Trott et Martinez Arias, 2013). Ces trois facteurs de transcription sont impliqués dans l’établissement et le maintien de la pluripotence, non seulement dans le cellules lors du clivage mais aussi dans les cellules souches embryonnaires (ES) ou les cellules iPS en culture (Chambers et Tomlinson, 2009). Ce RRG régule la pluripotence en réprimant les gènes impliqués dans la différenciation et en activant d’autres gènes importants pour la pluripotence (Navarro et al., 2012; Thomson et al., 2011).
Le nom du gène Nanog provient de Tír na nÓg, qui désigne en gaélique la « Terre de l’éternelle jeunesse » de la mythologie celtique.
Oct4, qui est connu également sous le nom de POU5F1, est un facteur de transcription à homéodomaine de type POU. Il est exprimé dans les cellules totipotentes ou pluripotentes au cours du développement et aussi dans les cellules germinales qui ont la capacité de participer à la génération suivante (donc virtuellement totipotentes à condition que la fécondation se réalise). La présence de Oct4 est essentielle pour le maintien des capacités pluri- ou totipotentes des cellules qui l’expriment (Stadtfeld et al., 2008). Par exemple, la perte de l’expression de Oct4 dans les cellules ES provoque la perte de leur pluripotence et aboutit à leur différenciation en trophectoderme (Niwa et al., 2000).

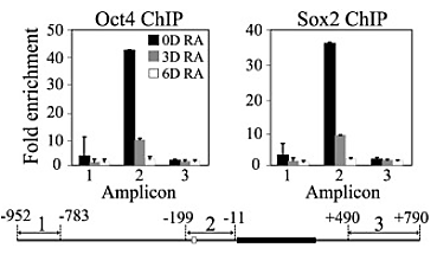
Sox2 contient un domaine HMG qui se lie au petit sillon de la double hélice d’ADN, tandis que Oct4 abrite un domaine POU qui interagit avec le grand sillon de l’ADN, permettant la formation de complexes ternaires Sox2-Oct4-ADN avec la paire de facteurs de transcription se liant à des motifs adjacents sur la séquence d’ADN et aussi se liant entre eux (Reményi et al., 2003; Williams et al., 2004). Des expériences d’immunoprécipitation de la chromatine suivie de séquençage (ChIP-seq) ont révélé que Sox2 et Oct4 occupent côte à côte des éléments régulateurs d’un grand nombre de gènes cibles et peuvent réguler leur expression de manière concertée (Li et al., 2019). Parmi ces cibles se trouve Nanog.
L’abaissement des niveaux d’expression de Nanog dans les cellules ES déclenche la différenciation et sa surexpression est suffisante pour maintenir les cellules dans un état pluripotent indépendant du LIF, une cytokine de la famille de l’interleukine-6 qui est ajouté habituellement dans le milieu de culture des cellules ES pour empêcher leur différenciation (Chambers et al., 2007).
La pluripotence des cellules dépend aussi de la traduction. En effet, plusieurs facteurs impliqués dans la maturation de la petite sous-unité ribosomique 40S sont préférentiellement exprimés dans les cellules souches pluripotentes par rapport à leur descendance différenciée et soutiennent la traduction de facteurs de transcription clés de la pluripotence tels que NANOG (You et al., 2015).
Pour tester la pluripotence de cellules souches, on peut les greffer sur des souris immunodéprimées (pour ne pas que la greffe soit rejetée). Les signaux qui maintiennent leur pluripotence sont levés et on obtient des tératomes contenant chacun de multiples lignages cellulaires. Egalement, on peut mettre les cellules en culture dans un milieu sans additifs maintenant leur état indéterminé et sans substrat auquel elles peuvent s’attacher : il se forme alors des agrégats appelés corps embryonnaires et où des cellules de différents lignages sont déterminées puis différenciées.

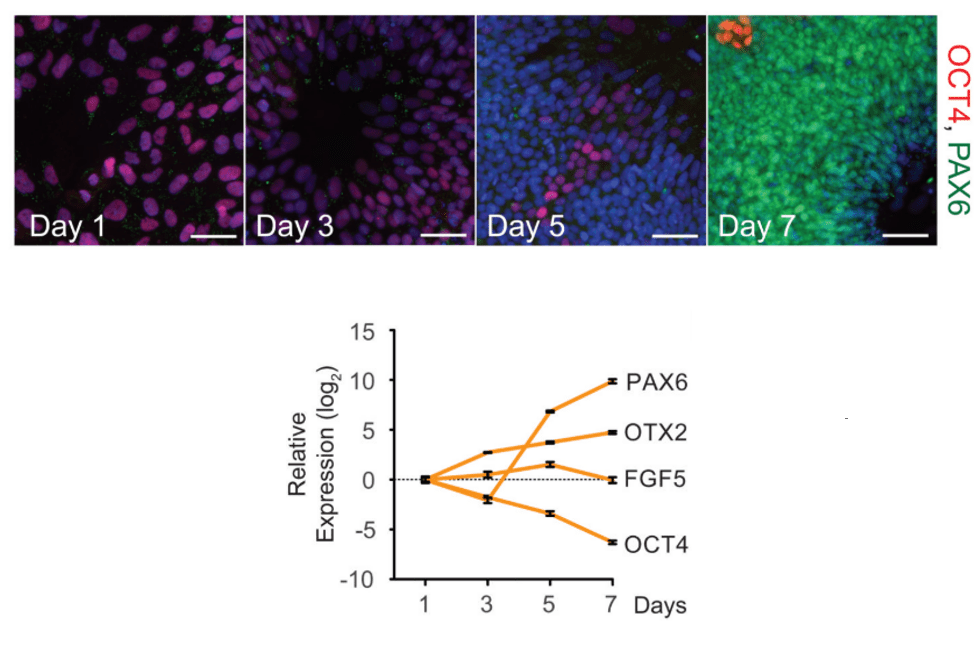
Signalons que Oct4 peut avoir un rôle plus ambigu : lors de la première étape de la sortie de l’état de pluripotence, il agit en concertation avec le facteur de transcription Otx2 et contribue à activer des gènes cibles qui font sortir les cellules de leur état naïf (Yang et al., 2014).
La pluripotence peut en effet être qualifiée de naïve comme ce que l’on observe dans la masse cellulaire interne et les cellules ES. Mais la pluripotence peut aussi être « primed » (primée ou avancée) comme dans les cellules de l’épiblaste post-implantatoire ou les cellules EpiSC. Cet état indique que les cellules s’avancent vers la détermination en des lignages spécifiques et notamment vers la détermination en feuillets (ectoderme, mésoderme, endoderme). Cela est lié notamment à des modifications de la chromatine (ADN plus méthylé dans un état primé que dans un état naïf) et à des modifications métaboliques (Weinberger et al., 2016; de Souza et al., 2025). Les facteurs de transcription Oct4, Sox2 et Nanog sont toujours actifs dans les cellules pluripotentes primées mais ils n’ont pas les mêmes cibles que dans les cellules pluripotentes naïves (Galonska et al., 2015).
Le retour à la pluripotence : le cas des cellules iPS
Les cellules souches pluripotentes induites iPS sont générées à partir de cellules différenciées. Cela implique de remonter au sommet dans le diagramme de Waddington. Le fait que mis dans un environnement favorable, un génome de cellules différenciées puisse fonctionner dans une cellule redevenu pluripotente était d’une certaine manière prédit par les expériences de clonage de John Gurdon vus plus haut. Il n’est donc pas étonnant que le Prix Nobel de Médecine de 2012 a été attribué conjointement à John Gurdon et à Shinya Yamanaka qui a mis au point la technique pour obtenir des cellules iPS.
Les cellules iPS sont obtenues en faisant surexprimer une combinaison de facteurs de transcription : Oct4, Sox2 que nous avons déjà évoqués et aussi Klf4 et c-Myc (facteurs OKMS ou facteurs Yamanaka). Les vecteurs d’expression de ces gènes sont introduits dans les cellules par des rétrovirus (la méthode la plus utilisée mais qui pose problème en thérapie cellulaire à cause de l’intégration des transgènes dans le genome), des adénovirus ou des épisomes (Wang et al., 2019).

Trois de ces quatre facteurs (Oct4, Sox2 et Klf4) sont considérés comme des facteurs de transcription pionniers, c’est-à-dire qu’ils peuvent se fixer sur des séquences cibles sur de l’ADN même au sein d’un nucléosome et sont capables d’outrepasser une forme répressive de la chromatine (Koche et al., 2011; Soufi et al., 2015). La fixation de ces trois facteurs facilite la fixation de c-Myc qui peut alors agir.

Les profils épigénétiques changent au cours de la production des cellules iPS avec un profil de méthylation et d’acétylation qui ouvre la chromatine (Krishnakumar et al., 2013). Par exemple, Klf4 recrute l’histone acetyltransférase p300 (Dai et al., 2014). Il y a un effacement de la « mémoire épigénétique » de la cellule différenciée. Cet effacement n’est toutefois pas complet comme le montre les variabilités entre lignées de cellules iPS provenant du même patient donc indépendante du génotype.
Lors de l’établissement des cellules iPS, il est clair que l’intérêt n’est pas tant de remonter le diagramme de Waddington que de pouvoir le redescendre ensuite et de générer un type cellulaire différent de celui des cellules qui ont été prélevées initialement. Il existe maintenant de très nombreux protocoles qui permettent d’activer les RRG souhaités pour aboutir aux cellules différenciées d’intérêt.
L’un des premiers effets de l’application des protocoles de différenciation aux cellules iPS est bien entendu l’extinction de l’expression des gènes du réseau de régulation génique de la pluripotence comme OCT4 ou NANOG.
Le potentiel thérapeutique des iPS est ainsi important.

En 2014, une équipe japonaise avait publié un article retentissant dans Nature concernant l’obtention de cellules souches pluripotentes appelées STAPS (pour Stimulus-Triggered Acquisition of Pluripotency). L’article décrivait qu’on pouvait obtenir ces cellules à partir de lymphocytes extraits de la rate en les soumettant simplement à une solution acide à pH5,7 pendant 25 minutes. Plusieurs laboratoires ont essayé de reproduire ces résultats, sans succès. Il s’est avéré qu’il s’agissait d’une fraude commise par une post-doctorante, première auteur de l’article qui a été rétracté. Les images des cellules exprimant des marqueurs de pluripotence provenaient en fait d’une autre expérience avec des cellules obtenues de manière plus classique.
Des cas de retour à de la multipotence
Il existe des cas « naturels » (et non plus artificiels comme la formation des cellules iPS) où des cellules regagnent de la multipotence et remontent dans le diagramme de Waddington. C’est le cas des cellules de crêtes neurales céphaliques qui, issues de l’ectoderme, sont capables de donner des dérivés mésodermiques comme du cartilage et de l’os. Elles réactivent pour ce faire transitoirement l’expression du gène de pluripotence Oct4 (Zalc et al., 2021). Cependant, elles ne donnent pas de dérivés endodermiques donc le terme de multipotence est celui qui s’applique.

Dans un autre système, après ablation des cellules souches de l’épithelium respiratoire, des cellules différenciées sont capables de se dédifférenciées en cellules souches multipotentes (Tata et al., 2014). De même, l’ablation des cellules souches intestinales multipotentes aboutit à leur restauration à partir de précurseurs déterminés en entérocytes ou en cellules sécrétrices (et même plus rarement à partir des cellules de Paneth (Mei et al., 2020). Cette plasticité est probablement expliquée par le fait que les progéniteurs des cellules sécrétrices et des entérocytes ont des niveaux comparables de marques d’histones H3K4me2 et H3K27ac et d’hypersensibilité à la DNaseI (signifiant une chromatine accessible et permissive) à la plupart des mêmes éléments régulateurs. Les enhancers agissant de manière spécifique dans les progéniteurs ont déjà des marques chromatiniennes d’activation dans les cellules souches intestinales révélant un amorçage épigénétique précoce pour des programmes transcriptionnels ultérieurement divergents (Kim et al., 2014).
Lors de l’amputation d’une partie du membre chiridien d’un axolotl, la régénération se fait grâce à un manchon de cellules dédifférenciées appelées blastème. Les souches d’axolotl transgéniques dans lesquelles les descendants de types cellulaires adultes distincts peuvent être marqués, suivis et isolés au cours du processus de régénération permettent de comprendre comment des lignées cellulaires particulières se développent pendant la formation du blastème et la régénération ultérieure des membres. La combinaison de ces souches d’axolotl transgéniques avec le séquençage d’ARN en cellules isolées (scRNA-seq) permet le suivi de types cellulaires individuels, ainsi que la reconstruction des étapes moléculaires (Gerber et al., 2018). Les cellules du tissu conjonctif, descendantes du mésoderme des lames latérales, sont la lignée la plus abondante contribuant au blastème (40% environ) et régénèrent les os et le cartilage, les tendons, le périsquelette et les fibroblastes dermiques et interstitiels. Les restrictions de lignage sont cependant plus marquées pour les autres cas et ne passent pas des cellules multipotentes : les nouvelles cellules musculaires proviennent seulement de cellules musculaires, les nouvelles cellules de Schwann proviennent uniquement des cellules de Schwann et ainsi de suite.
De la pluripotence à la totipotence
Les cellules 2CLC (2-cell-like cells) sont des cellules dérivées de cellules souches embryonnaires (ES) qui exhibent des caractéristiques transcriptionnelles, chromatiniennes et métaboliques similaires aux blastomères du stade embryonnaire à 2 cellules chez la souris. L’intérêt majeur des 2CLCs réside dans leur état similaire à la totipotence, c’est-à-dire qu’elles possèdent la capacité d’engendrer à la fois des tissus embryonnaires et extraembryonnaires, ce qui dépasse la simple pluripotence des cellules ES classiques. Les 2CLCs offrent ainsi un modèle précieux pour étudier les mécanismes du développement très précoce : activation du génome zygotique, plasticité cellulaire au tout début du développement embryonnaire, et processus épigénétiques liés à la totipotence (Jia et al., 2024; Zhu et al., 2024). Il a ainsi été démontré grâce à ce modèle que les cellules totipotentes répliquent leur ADN plus lentement que des cellules avec des potentialités plus restreintes. Si on ralentit la réplication dans des cellules ES, cela induit la reprogrammation en cellules 2CLC (Nakatani et al., 2022). Cela est du au fait que durant la réplication des changements majeurs dans les marqueurs épigénétiques peuvent avoir lieu.
OUTILS :
BioTapestry : un logiciel gratuit pour créer des représentation de réseaux de régulation génique.
EN DIRECT DES LABORATOIRES :
Geneviève Almouzni -Institut Curie :
QUELQUES EQUIPES DE RECHERCHE FRANCOPHONES DANS LE DOMAINE :
Embryon et pluripotence : épigénétique et environnement (EPEE) – INRAE, Jouy-en-Josas
- Adhérences cellule-cellule
- Arabidopsis thaliana
- Axe antéro-postérieur chez la drosophile
- Biomécanique du développement
- Caenorhabditis elegans
- Concepts principaux
- Contrôle de la traduction
- Contrôle de la transcription
- Contrôle génétique et épigénétique
- Croissance du tube pollinique et double fécondation chez les Angiospermes
- Croissance et guidage axonal
- Des modèles animaux moins classiques
- Développement de l’oeil des Vertébrés
- Développement et évolution
- Et l’Humain ?
- Exercices sur l’ovogenèse, la spermatogenèse et la fécondation
- Exercices sur le contrôle de l’expression des gènes
- Exercices sur le développement des bourgeons de membre
- Exercices sur le développement des muscles striés squelettiques
- Exercices sur le développement des végétaux et les hormones végétales
- Exercices sur les cycles et les divisions cellulaires
- Exercices sur les étapes du développement, les inductions embryonnaires et la mise en place des axes de polarité
- Exercices sur les matrices extracellulaires, le cytosquelette et les adhérences cellule-cellule
- Exercices sur les voies de signalisation
- Glossaire
- Glossaire des termes liés à la génétique
- Glossaire des termes liés au cytosquelette, la matrice extracellulaire, l’adhérence et la migration cellulaire
- Hématopoïèse et développement des cellules du système immunitaire
- Histoire de la biologie cellulaire et de la biologie du développement
- L’acide rétinoïque
- L’apoptose
- L’autophagie
- L’organogenèse
- L’ovogénèse prépare le développement embryonnaire
- La drosophile
- La fécondation
- La formation des somites
- La gastrulation
- La gastrulation (version allégée)
- La métamorphose chez les Hexapodes et les Amphibiens
- La neurogénèse chez les mammifères adultes
- La neurulation
- La poule
- La signalisation calcique
- La souris
- La superfamille TGFβ et ses voies de signalisation
- La voie de signalisation de l’auxine et ses rôles
- La voie de signalisation Hedgehog
- La voie de signalisation Hippo et ses composants YAP/TAZ
- La voie de signalisation Notch
- Le clivage
- Le cytosquelette
- Le développement des bourgeons de membre
- Le développement des muscles striés squelettiques
- Le développement des organes génitaux et des cellules germinales
- Le développement du cortex
- Le méristème apical caulinaire en phase végétative et lors de la formation d’une fleur
- Le poisson zèbre
- Le xénope
- Les cellules des crêtes neurales
- Les cellules et les gènes en action dans le développement
- Les cellules souches
- Les cellules tumorales
- Les cycles et les divisions cellulaires
- Les étapes du développement
- Les étapes du développement embryonnaire d’Arabidopsis thaliana et leur contrôle
- Les inductions embryonnaires et les gradients de morphogène
- Les matrices extracellulaires animales
- Les organismes modèles
- Les outils pour étudier l’expression et la fonction des gènes
- Les parois des cellules végétales
- Les techniques et les outils pour la biologie cellulaire
- Les transitions épithélio-mésenchymateuses et les migrations cellulaires
- Les vésicules extracellulaires
- Les voies de signalisation
- Les voies de signalisation FGF
- Mise en place des axes chez les Vertébrés
- Structures et processus cellulaires
- Voies de signalisation WNT