par Patrick PLA, Université Paris-Saclay
SOMMAIRE :
- Introduction
- Initiation de la traduction
- Translocation et terminaison de la traduction
- Contrôle de la dégradation des ARNm
- Contrôle de la localisation de la traduction
- Les microARN et l’inhibition de la traduction
Introduction
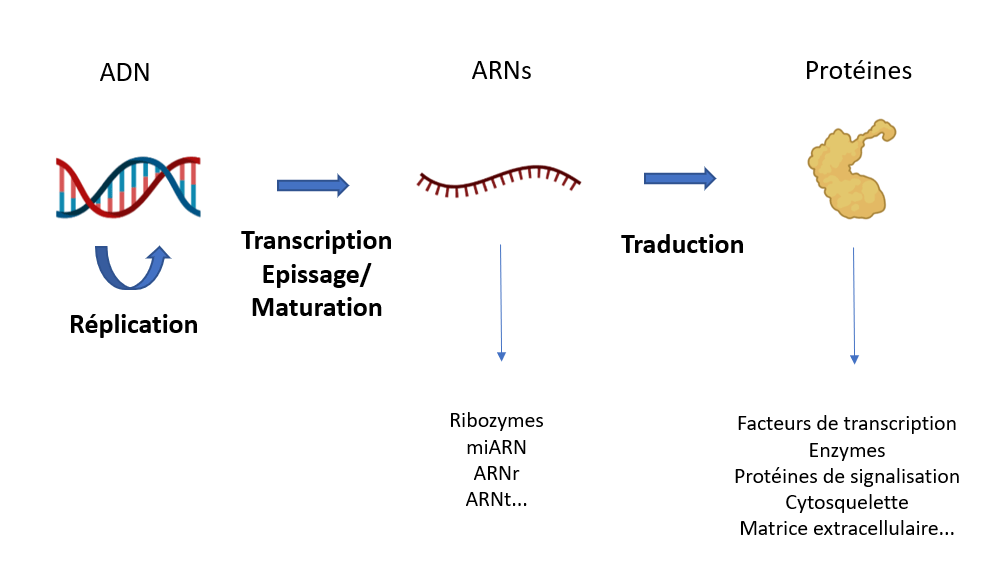
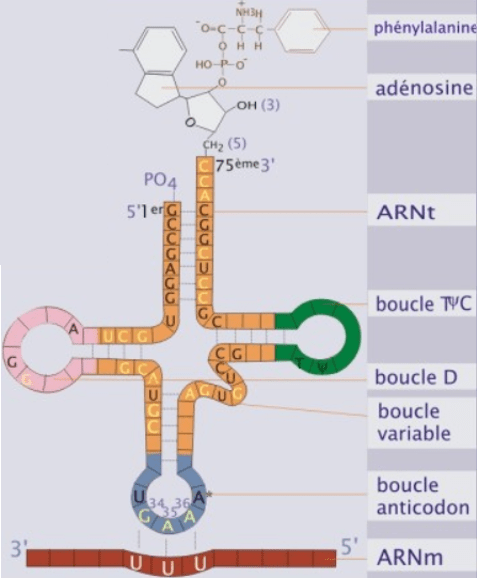
La conversion de l’information portée par l’ARNm en protéine représente la traduction. Il s’agit de l’interprétation de la séquence des ribonucléotides portée par l’ARNm en acides aminés. Elle est réalisée selon un code appelé le code génétique. Ce sont les ribosomes, des complexes formés de protéines et d’ARNr (ARN ribosomique) qui réalisent la traduction avec l’aide des ARNt (ARN de transfert).

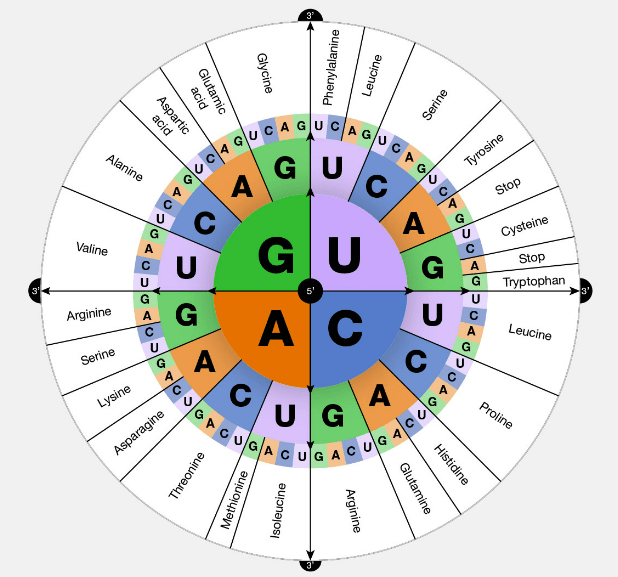
Dans ce système, un triplet de nucléotides, ou codon, désigne un acide aminé. Les ARNt permettent de faire le couplage entre un codon donné et l’acide aminé correspondant. Leur séquence comporte une séquence complémentaire au codon : l’anti-codon. Il y a quatre codons particuliers. Un codon AUG appelé codon-initiateur codant pour la méthionine permet de commencer la traduction et 3 codons appelés codon stop (UAA, UAG, UGA) provoquent l’arrêt de la traduction.
Cependant, la complémentarité de bases Watson-Crick n’est pas toujours parfaite en ce qui concerne le troisième nucléotide du codon et le nucléotide de l’anti-codon correspondant sur l’ARNt. Notamment, certains ARNt ont pour base azotée à cet endroit de l’anti-codon l’hypoxanthine (au sein d’un nucléoside appelé inosine). Cette base azotée peut s’associer avec un A, un C et un U. Dans d’autres cas, le G de l’anti-codon peut s’associer au U d’un codon en plus du C habituel. Tous ces cas d’appariement de bases imprécis sont appelés wobble (qui veut dire bancal en anglais).
La traduction s’effectue dans le cytoplasme, mais sa localisation précise peut être contrôlée (soit par la localisation de l’ARNm, soit par la localisation des ribosomes).
Initiation de la traduction
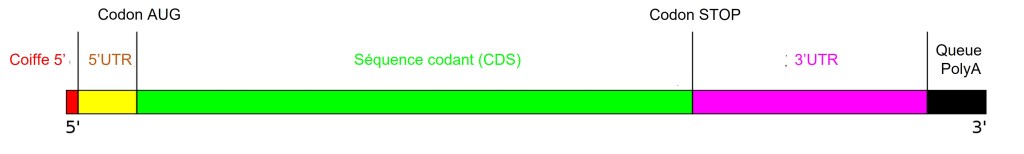
Lorsque l’ARNm arrive dans le cytosol, il est pris en charge par des facteurs d’initiation de la traduction. Ces protéines éliminent la protéine de coiffe (protéine CBC) puis interagissent avec les protéines fixées sur la queue poly A. Ainsi, la traduction ne peut démarrer que si les deux extrémités de l’ARNm sont intactes.
Parmi les facteurs d’initiation de la traduction, citons eIF4E qui se lie à la coiffe 5′ des ARNm. Son activité peut être augmentée par une phosphorylation sur la sérine 209 à la suite de la réception par la cellule de facteurs de croissance.

eIF4E est surexprimé dans une grande variété de tumeurs, notamment les mélanomes. On pense qu’il stimule la tumorigenèse et la progression tumorale en facilitant la traduction des ARNm qui codent des protéines stimulant la prolifération et la survie. Dans les mélanomes métastatiques, une forte phosphorylation de eIF4E sur la sérine 209 est associé avec un mauvais pronostic (Carter et al., 2016).
eIF4E forme le complexe eIF4F avec la protéine d’échafaudage eIF4G et une hélicase eIF4A pour initier la traduction dépendante de la coiffe.
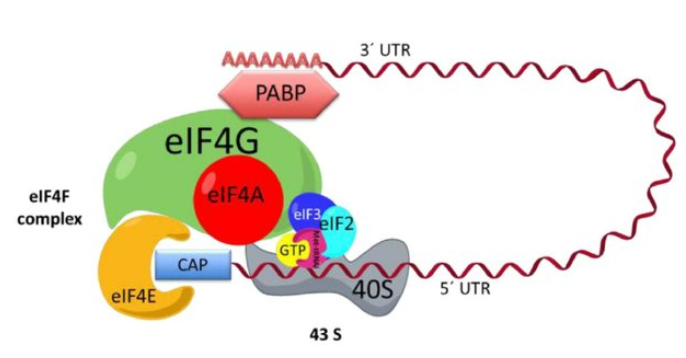
L’activité des facteurs d’initiation de la traduction peut être modulée par des phosphorylations. Par exemple, dans une cellules végétale en cas de pénurie d’acides aminés, la kinase GCN2 phosphoryle eIF2 ce qui la rend moins active et l’activité traductionnelle est globalement diminuée (Lageix et al., 2008).
La traduction est ensuite initiée par le complexe dit 43S qui contient la petite sous-unité du ribosome (40S) qui se fixe sur l’extrémité 5’ de l’ARNm, aidée par des facteurs d’initiation de la traduction qui ont reconnu la coiffe 5′. Cette petite sous-unité du ribosome est accompagnée de l’ARNt initiateur qui porte la méthionine. La sous-unité se déplace jusqu’à ce qu’elle parvienne au codon-initiateur. Alors, il y a un changement de conformation du complexe et les facteurs d’initiation se dissocient et la grande sous-unité ribosomique (60S) se fixe dans une position permettant à l’ARNt initiateur d’occuper le site P du ribosome. Ensuite, un second ARNt chargé de son acide aminé et accompagné d’un facteur d’élongation associé au GTP, vient occuper le site A du ribosome.
Dans certains cas de stress (hypoxie, infection virale, génotoxicité), la cellule diminue le niveau global de traduction en freinant son initiation. La traduction habituelle dépendante de la coiffe est compromise car eIF4E est lié et séquestré par la protéine 4E-BP1 (Richter et Sonenberg, 2005). Cependant, en cas d’hypoxie par exemple, on observe que la traduction des ARNm codant des facteurs de croissance angiogéniques (comme le VEGF) qui permettent d’initier la formation de nouveaux vaisseaux sanguins est stimulée. Le blocage général de la traduction est surmonté par l’utilisation comme lieu d’assemblage des ribosomes non pas de la coiffe 5′ mais d’un IRES (pour internal ribosome entry site). L’IRES se trouve dans la région 5′ non traduite (5’UTR) des ARNm. Le complexe d’initiation est alors composé d’ITAF (IRES trans-acting factors) et de longs ARN non codant comme long ARN non codant comme NEAT1 (nuclear-enriched abundant transcript 1) (Godet et al., 2022). Dans certains cas, ce complexe s’associe à l’ARNm dès le noyau (la traduction a toujours lieu dans le cytoplasme).
Translocation et terminaison de la traduction
La translocation est le déplacement relatif de l’ARNm et du ribosome. Elle permet la croissance de la chaine polypeptidique de 7 à 15 acides aminés par seconde chez les Procaryotes et de 3 à 5 acides aminés par seconde chez les Eucaryotes.
L’appariement codon-anticodon est le facteur essentiel de sélection. Si l’appariement codon-anticodon est suffisamment fort, le GTP est hydrolysé ce qui provoque la dissociation du facteur d’élongation. L’hydrolyse du GTP produit également un changement de conformation du ribosome, ce qui active l’ARNr 28S (ARNr 25S chez les plantes). Il s’agit d’un ribozyme qui catalyse la formation d’une liaison peptidique entre la méthionine et le deuxième acide aminé. Si l’appariement codon-anticodon n’est pas suffisamment fort, l’ARNt quitte le site A sans que le GTP puisse être hydrolysé et donc la liaison entre les acides aminés n’est pas réalisée. Des essais successifs de différents couples ARNt-acide aminé qui se présentent de façon aléatoire peuvent se succéder. Une fois la liaison peptidique réalisée, l’hydrolyse du GTP lié à un second facteur d’élongation provoque un second changement de conformation du ribosome menant à la translocation. Le site A se trouve libre pour recevoir un troisième ARNt chargé d’un troisième acide aminé et ainsi de suite.
Jusqu’à une période récente, on pensait que les codons synonymes (ceux qui présentent des séquences différentes sur l’ARNm mais qui aboutissent à un même acide aminé) ne provoquaient pas de différences et avaient été négligés dans l’étude des mutations humaines par exemple. On s’est rendu compte que certains codons sont utilisés plus rarement que d’autres et que la quantité d’ARNt correspondant à ces codons peuvent être rares, ralentissant la translocation et donc la production de la chaîne polypeptidique. Cela peut entraîner des conséquences sur la mise en place de la conformation des protéines et altérer leur fonction (Walsh et al., 2020).
La vitesse à laquelle se fait la translocation et l’élongation de la chaine polypeptidique en croissance est variable selon la séquence qui est lue mais aussi selon l’âge de l’organisme. Des pauses de la traduction peuvent même s’observer ce qui peut être bénéfique pour laisser le temps à des domaines de la protéine de prendre leur bonne conformation avec éventuellement l’aide de protéines chaperons (Kim et al., 2015). Mais avec l’âge, des pauses traductionnelles plus longues sont observées chez la levure et chez C. elegans aboutissant à des « embouteillages de ribosomes » et à des protéines naissantes avec des conformations partielles qui peuvent former des agrégats (Choe et al., 2016; Stein et al., 2022). De tels processus ont été impliqués dans la neurodégénération (Ishimura et al., 2014).
Pendant la phase d’élongation, les ribosomes peuvent se bloquer trop longtemps lorsqu’il y a des déficits en ARNt, des défauts de décodage, une obstruction par des structures secondaires de l’ARN, des dommages chimiques à l’ARN ou des séquences peptidiques naissantes inhibitrices (Schuller et Green, 2018). Il arrive alors que le ribosome suiveur rattrape et entre en collision avec le ribosome principal bloqué, formant une structure dimérique ribosomique inhabituelle appelée disome (ou trisome si trois ribosomes entrent en collision). La machinerie cellulaire reconnaît les disomes et le ribosome bloqué peut être dégradé tout comme le polypeptide généré incomplet via la voie du contrôle qualité associé au ribosome (RQC) (Ikeuchi et al., 2019; Sitron et Brandman, 2020). De plus, la dégradation no-go (NGD) dégrade l’ARNm sur lequel les ribosomes se bloquent, empêchant ainsi l’accumulation d’ARNm défectueux (Simms et al., 2017). C’est la protéine Hel2 qui reconnait les disomes et agit comme une E3 ligase et modifie les protéines ribosomales par ubiquitination (Ikeuchi et al., 2019)
Une chaîne polypeptidique naissante en cours d’élongation interagit avec l’intérieur d’un tunnel ribosomal étroit avant de sortir dans le cytosol, où elle continue de subir l’influence du ribosome par le biais d’interactions transitoires avec sa surface externe (Voss et al., 2006; Ellis et al., 2008). Le potentiel électrostatique négatif du ribosome, provenant de ses ARN ainsi que des résidus exposés à la surface de ses protéines, chargés négativement, peut moduler les interactions avec la chaîne polypeptidique en croissance. Ces interactions peuvent influencer les processus de repliement cotraductionnel, probablement en stabilisant les conformations désordonnées prédominantes dans les chaînes polypeptidiques incomplètes, et peuvent même retarder la formation de la structure native et l’assemblage moléculaire des complexes protéiques (Kaiser et al., 2011; Cabrita et al., 2016; Marino et al., 2018). Une fois la nouvelle chaîne polypeptidique suffisamment sortie, son repliement peut également être assisté par des protéines chaperones (Hoffmann et al., 2012).
Les translocations se poursuivent avec ajout d’acides aminés à la chaîne polypeptidique en cours de synthèse jusqu’à la terminaison. Celle-ci est programmée dans l’ARN messager par un des trois codons stop, qui est lié à un facteur de terminaison de l’élongation associé au GTP. Aucun ARNt possède l’anticodon correspondant, le site A reste donc inoccupé. L’hydrolyse de la liaison du dernier acide aminé avec son ARNt signe la fin de la traduction. Il se forme ainsi l’extrémité carboxy terminale de la protéine. Les deux sous-unités du ribosome se dissocient et libèrent l’ARNm qui peut être traduit à nouveau (phénomène d’amplification : un gène peut être transcrit de multiples fois en ARNm et chaque ARNm peut être traduit de multiples fois).
Récemment, il est apparu que les ARNt n’étaient pas de simples liens entre les codons et les acides aminés. Les ARNt peuvent être clivés pour former des 5’tRFI ou des 3’tRFI selon le fragment considéré et ils peuvent inhiber ou faciliter la traduction selon le contexte, parfois associés aux protéine de type Argonaute, de manière proche aux microARN (Martinez et al., 2017). Ils peuvent même contrôler la transcription d’après un mécanisme qui commence à être élucidé (Boskovic et al., 2019). Les 5’tRFIGly/GCC (c’est-à-dire un fragment 5′ de l’ARNt lié à la glycine et qui reconnait le codon GCC) et 5’tRFIGlu/CTC sont capables d’activer la transcription de leur propre gène ce qui augmente la quantité d’ARNt disponible. Cela a lieu autour de la MBT (transition mi-blastuléenne) chez le poisson-zèbre et si on inhibe ce phénomène, le développement est anormal. Il s’agit d’une boucle de rétroaction positive qui doit augmenter la production d’ARNt pour permettre de traduire les nouveaux ARNm à la reprise de la transcription lors de la MBT et pour pouvoir produire une quantité importante de nouvelles protéines (Chen et al., 2021).
Dans le cytoplasme, la région 3’ non codante des ARNm permet de contrôler l’amorçage de la traduction. Ce contrôle dépend de modifications de la polyadénylation des ARNm qui doivent être stockés dans les cellules. C’est particulièrement le cas dans les ovocytes où des ARNm sont stockés dans le cytoplasme au cours de l’ovogénèse et seront indispensables aux premiers stades du développement où il n’y a pas de transcription (ces ARNm sont codés par des gènes dits à effet maternel). La traduction de ces ARNm stockés doit être temporairement réprimée, jusqu’à être activée au bon moment après la fécondation. Les ovocytes contiennent de nombreux ARNm de réserve dont la queue polyA est réduite à 20-40 résidus. Dans ces conditions, les protéines de liaison à l’extrémité 3’ ne se fixent pas, mais une protéine, la maskine, reconnaît la courte queue poly A, s’y fixe et interagit avec la coiffe en 5’. Elle empêche alors l’interaction de la coiffe avec les facteurs d’initiation de la traduction et la fixation de la petite sous-unité des ribosomes. Si l’ovulation ou la fécondation ont lieu, la maskine est déplacée, ce qui permet à la poly A-polymérase d’allonger la queue polyA. Les protéines qui participent à la traduction peuvent alors se fixer et favoriser le recrutement des ribosomes.
Focus sur les ribosomopathies
Les ribosomopathies sont des maladies caractérisées par des mutations dans les composants du ribosome ou des facteurs clés de la biogenèse du ribosome qui produisent des phénotypes spécifiques dans divers tissus. Par exemple, l’anémie Diamond Blackfan (DBA), qui résulte d’une haploinsuffisance de ∼20 différentes protéines ribosomiques essentielles (RP), a pour symptômes des anomalies congénitales mal comprises, telles que des malformations craniofaciales et des membres (notamment des doigts), ainsi qu’une altération de la différenciation érythroïde (Sieff, 2010; Yelick et Trainor, 2015). Le défi scientifique posé par cette maladie est de comprendre comment ce qui affecte des protéines qui sont exprimées presque partout puissent avoir des effets aussi spécifiques.
De nombreuses études suggèrent que les phénotypes des ribosomopathies peuvent résulter de l’activation du facteur de transcription p53 induit par le stress. En particulier, on pense que des mutations dans les protéines ribosomiques conduisent à la stabilisation et à l’activation de p53 par un mécanisme dans lequel le stress nucléolaire et la biogenèse de ribosomes défectueux entraîne l’inhibition du principal régulateur inhibiteur de p53, Mdm2 (Deisenroth et al., 2010; Dutt et al., 2011, Morgado-Palacin et al., 2015). La stabilisation de p53 contribue aux ribosomopathies via l’activation transcriptionnelle de gènes impliqués dans la régulation de la prolifération cellulaire et de l’apoptose (Deisenroth et Zhang, 2010). Le rôle de p53 dans la génération de ces phénotypes est bien mis en évidence lors de l’examen de l’haploinsuffisance de RP in vivo. Des études sur des modèles de souris et de poisson zèbre de DBA ont démontré que l’haploinsuffisance de RP peut entraîner des défauts spécifiques aux tissus, qui peuvent être sauvés par la perte de p53 (Barlow et al., 2010; Danilova et al., 2008; McGowan et Mason, 2011). Bien que ces résultats mettent en évidence le rôle de p53 dans le développement de phénotypes de maladies, la façon dont les effets globaux médiés par p53 sur les cellules, tels que l’arrêt du cycle cellulaire et l’apoptose, peuvent conduire à des phénotypes de développement aussi spécifiques aux tissus reste un paradoxe.
Alternativement, il a été suggéré que des mutations dans les RP peuvent conduire à une dérégulation traductionnelle indépendante de p53 (Dalla Venezia et al., 2019; Khajuria et al., 2018). Malgré cela, les modifications apportées à la traduction ont été largement inexplorées dans les contextes de développement in vivo associés à l’haploinsuffisance RP. Une récente étude de culture cellulaire a identifié des défauts de traduction dans les cellules hématopoïétiques lors de la déplétion des RP habituellement mutées dans le DBA et suggère que les niveaux de ribosomes peuvent jouer un rôle dans ce dysfonctionnement (Khajuria et al., 2018). Cependant, il est difficile de savoir comment cette hypothèse rend compte de phénotypes complexes in vivo et comment elle peut être conciliée avec le rôle connu de p53 dans l’étiologie des phénotypes de ribosomopathie.
Il existe de plus en plus de preuves que le ribosome peut se diversifier structurellement et fonctionnellement pour réguler la traduction (Gilbert, 2011 ; Sulima et Dinman, 2019). Par exemple, l’expression spécifique de la grande protéine de sous-unité ribosomique L38 (RPL38) (Kondrashov et al., 2011) et les segments d’expansion ribosomique (Leppek et al., 2020) régulent la traduction de certains gènes Hox.
Les paralogues des protéines ribosomiques (RP) diversifient l’activité des ribosomes au cours du développement des gonades (Hopes et al., 2021), et l’hétérogénéité intracellulaire des ribosomes régule la traduction (Shi et al., 2017).
Des protéines a priori non identifiées comme ribosomiques peuvent s’associer aux ribosomes dans certaines circonstances, comme SOX2, un facteur surtout connu pour réguler la transcription dans les cellules souches. La partie C-terminale de SOX2 est capable de s’associer aux ribosomes et cela modifie l’efficacité de la traduction d’un certain nombre d’ARNm dont le produit est impliqué dans des fonctions importantes comme les interactions avec la matrice extracellulaire ou les voies de signalisation (Schaefer et al., 2024).
Les segments d’expansion (ES) consistent en une multitude de structures d’ARNr qui ressemblent à des tentacules et qui s’étendent à partir du ribosome central. Les ES sont remarquablement variables en séquence et en taille à travers l’évolution eucaryote avec des fonctions largement inconnues. En caractérisant la liaison du ribosome à un élément régulateur dans le 5′ UTR de l’ARNm de Hoxa9, des chercheurs ont identifié une structure secondaire sous la forme d’une tige-boucle modulaire qui se lie à un seul ES, ES9S. L’édition du génome pour perturber spécifiquement le site de l’interaction Hoxa9-ES9S aboutit à une production plus faible de la protéine HoxA9 ce qui démontre l’importance fonctionnelle d’une telle liaison sélective ARNm-ARNr dans le contrôle de la traduction.
Les modifications post-transcriptionnelles des ARNr comme la 2’-O-méthylation sur le ribose peuvent aussi contribuer à des modifications de l’efficacité de la traduction (Khoshnevis et al., 2022).
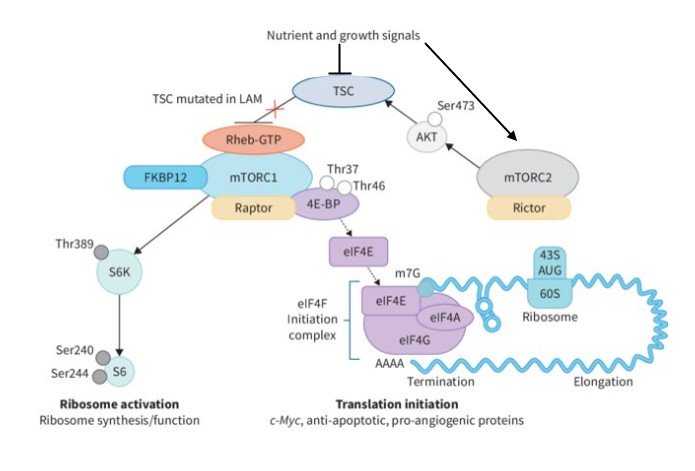
La kinase mTORC1 est un régulateur central de la synthèse des protéines et de la croissance cellulaire. Son activité est augmentée par la présence simultanée de facteurs de croissance et d’une bonne quantité de nutriments. mTORC1 phosphoryle plusieurs cibles et stimule les étapes d’initiation et d’élongation de la traduction de l’ARNm, aboutissant ainsi à une accélération de la production de nouvelles protéines.
Contrôle de la dégradation des ARNm
La déadénylation, c’est-à-dire le raccourcissement de la queue polyA, est une première étape de la dégradation des ARNm cytoplasmiques (Wiederhold et Passmore, 2010). En général, la queue polyA d’un ARNm est de 200 nucléotides environ. Si la longueur de la queue polyA tombe sous les 30 nucléotides, l’ARNm est le plus souvent dégradé. Cette dégradation a lieu le plus souvent dans les corps P (traduction de Processing bodies ou P bodies), des structures cytoplasmiques rassemblant de nombreuses protéines, tels que les complexes qui enlèvent la coiffe 5′ (DCP1, DCP2), l’exonucléase XRN1 (qui agit en 5’→3’), et le complexe de déadénylation (CCR4-NOT).

Le nombre de corps P dans les cellules peut varier, notamment augmenter en l’espace de quelques minutes lorsque la cellule est soumise à un stress (Riggs et al., 2020). Notez que certains ARNm non traduits peuvent être stockés dans les corps P et ressortir pour éventuellement être traduits plus tard (Courel et al., 2018).
Le complexe enzymatique impliqué dans le raccourcissement poly(A) est le complexe CCR4-NOT (Doidge et al., 2012; Temme et al., 2014). Chez les mammifères, l’importance physiologique de la déadénylation de l’ARNm médiée par le complexe CCR4-NOT a été démontrée dans les spermatocytes et les ovocytes en parallèle avec la méiose où la dégradation des ARNm est massive (Sha et al., 2018 ; Yu et al., 2016) puis ce complexe a été retrouvé agir dans de multiples types cellulaires (Mostafa et al., 2020).
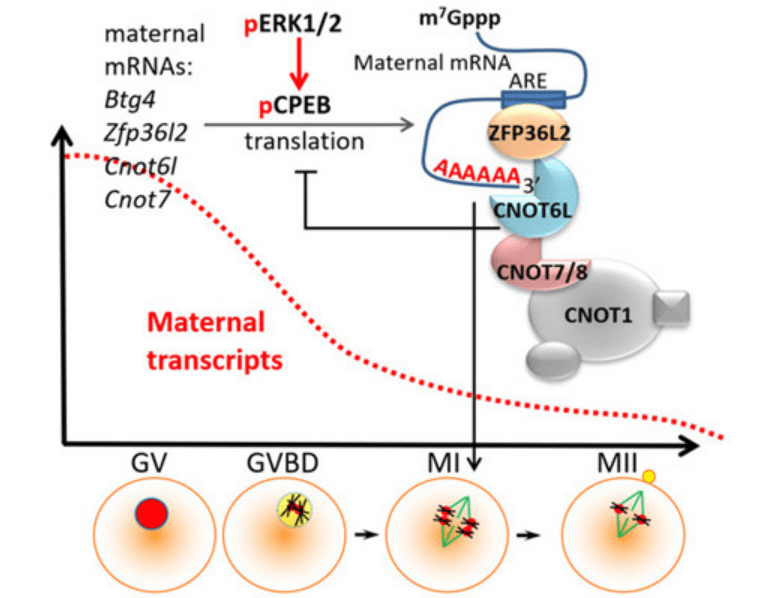
La dégradation des ARNm par déadénylation peut être provoquée par des microARN. Les microARN sont des oligonucléotides de 20 et 25 nucléotides produits par l’ARN polymérase II (la même qui synthétise les ARNm) et qui sont complémentaires d’une séquence souvent présente dans le 3’UTR de ces ARNm cibles. Certains d’entre eux peuvent provoquer une déadénylation qui aboutit à la dégradation de leurs ARNm cibles comme par exemple miR-430 qui est capable de cibler plusieurs centaines d’ARNm différents et qui est essentiel pour la dégradation des ARNm maternels lorsque la transcription des gènes de l’embryon du poisson-zèbre s’active, permettant d’accélérer la transition entre une phase où les gènes à effets maternels sont impliqués vers une phase où les gènes dits zygotiques sont exprimés (Giraldez et al., 2006).
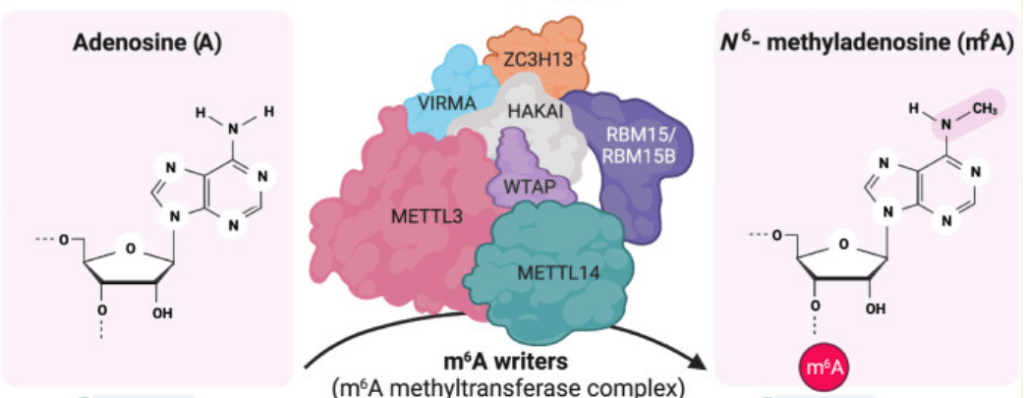
La dégradation des ARNm peut être provoquée par une méthylation de l’adénosine. Il s’agit d’une modification courante de l’ARN chez les eucaryotes. Le groupe méthyle est attaché à la position azote-6 de l’adénosine, créant la N6-méthyladénosine ou m6A. Cette modification est reconnue chez la drosophile par la protéine FMR1 au sein d’un motif AGACU sur les ARNm maternels et aboutit à la stimulation de leur dégradation (Zhang et al., 2022).
La dégradation peut être précédée de l’élimination de la coiffe 5′ ce qui rend les ARNm cibles incapables d’être traduits (Charenton et Graille, 2018). La déplétion chez la souris de la protéine qui réalise cette élimination, Dcp1a, aboutit à une léthalité à E10,5 avec un grand retard de développement, montrant le rôle fondamental de ce mécanisme (Ibayashi et al., 2021). Une fois la coiffe 5′ enlevée, l’exoribonucléase XRN1 dégrade l’ARNm dans la direction 5′ vers 3′.
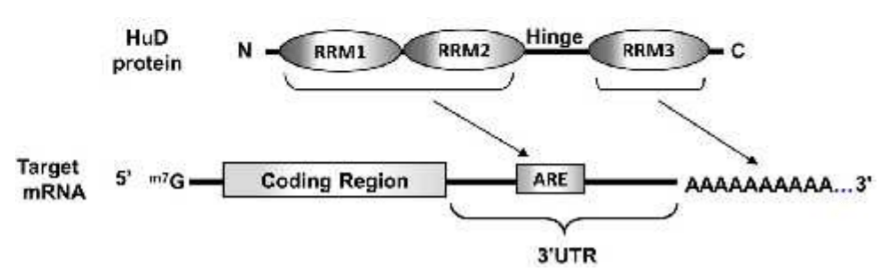
Des ARNm peuvent être protégés de la dégradation. Par exemple au cours du développement puis du fonctionnement chez l’adulte du système nerveux, des protéines se liant à l’ARN appelées HuA, HuB, HuC et HuD stabilisent deux familles d’ARNm qui, sans ces protéines, seraient dégradées plus vite (Perrone-Bizzozero et Bird, 2013). Des mutations dans HuD ont été associées à des déclenchements précoces de la maladie de Parkinson (Nourredine et al., 2005).
Contrôle de la localisation de la traduction
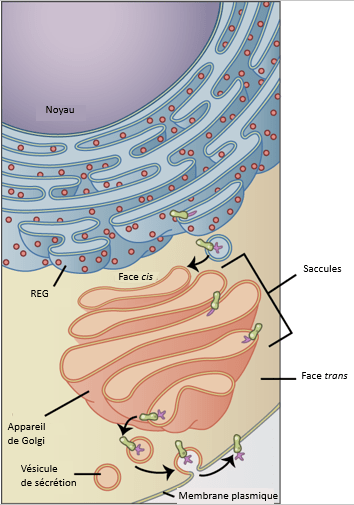
Il existe un premier contrôle de la localisation de la traduction via la reconnaissance du peptide signal en 5′ de la protéine naissante durant le début de la traduction. Ce peptide signal est essentiellement composé d’acides aminés hydrophobes et est reconnu par un complexe SRP (constitué par 6 protéines et un ARN de 300 nucléotides appelé 7S) qui arrime les ribosomes au reticulum endoplasmique qui devient granuleux (REG).

2) Le complexe SRP/ribosome se lie au récepteur SRP sur le reticulum endoplasmique de manière dépendante au GTP (les 2 pastilles rouges)
3) Le ribosome est ainsi arrimé proche du translocon (ou complexe de translocation du peptide) et l’allongement reprend. Le peptide signal reste dans le pore du translocon tandis que les régions solubles du polypeptide sont enfilées dans la lumière du RE sous forme de boucles. Le complexe SRP est libéré lorsqu’il y a lyse du GTP en GDP.
La protéine en production traverse alors la membrane du REG. Elle peut s’y arrêter et devenir transmembranaire ou alors poursuivre son passage et se retrouver dans la lumière du REG. Cela concerne notamment les protéines secrétées qui, lorsqu’elles sont des morphogènes ou font partie de la matrice extracellulaire, ont un rôle considérable au cours du développement.
Signalons que le peptide signal est clivé par une peptidase dans le REG et ne fait donc pas partie des protéines matures.
La localisation de la traduction au REG n’est pas seulement un problème d’adressage. Dans le système endomembranaire qui est constitué par le REG, l’appareil de Golgi et les vésicules de sécrétion, les protéines issues de la traduction peuvent subir une maturation et des modifications post-traductionnelles qui affectent leur fonction de manière essentielle. Parfois, des protéines chaperons les aident à avoir une conformation correcte. Par exemple, la protéine chaperon Mesd aide LPR5 et LRP6, les co-récepteurs des ligands Wnt, à avoir une conformation fonctionnelle. En son absence, les souris mutées présentent des défauts de développement de même nature qu’une déficience en signalisation Wnt (Hsieh et al., 2003).
Mais à l’intérieur même de la catégorie des protéines traduites dans le cytoplasme (et non pas à la surface du REG), il peut exister des localisations particulières de la traduction qui résultent de la localisation des ARNm dans des régions spécifiques de la cellule. Par exemple, une partie des ARNm codant l’agrine, une protéine jouant un rôle dans la formation des synapses neuro-musculaires est transportée vers l’extrémité de l’axone des motoneurones et l’agrine est alors synthétisée à proximité immédiate de la synapse (Tu et al., 2024).
Ce ciblage et cette localisation asymétrique de l’ARNm sont le résultat d’une cascade d’événements ordonnés qui consistent en la formation d’un complexe ribonucléoprotéique, le transport du complexe vers les sites d’ancrage, l’ancrage des particules au cytosquelette et la traduction locale des ARNm.
Dans la plupart des cas, la localisation des ARNm est spécifiée par des séquences de la région 3’ non traduite (3’UTR) des ARNm. C’est par exemple le cas dans les ovocytes de drosophile et d’amphibien, les myoblastes, les neurones et les fibroblastes. Dans ces deux derniers types de cellules, les ARNm de l’actine α et γ sont présents autour du noyau tandis que celui de l’actine β est localisé à la périphérie des cellules au niveau des lamellipodes (ou des cônes de croissance chez les jeunes neurones). L’ARNm codant cette forme d’actine possède des séquences particulières sur son 3’UTR qui permet ce transport vers le cortex cellulaire.
Dynamique en temps réel de l’ARNm de la β-actine dans une cellule en migration. La technique de marquage MS2-GFP a été utilisée pour visualiser l’ARNm endogène de la β-actine. La carte des couleurs montre l’intensité de fluorescence de NLS-MCP-GFP divisée par l’intensité de colorant cytoplasmique, représentant la concentration d’ARNm de β-actine (Hye Yoon Park and Robert H. Singer, Albert Einstein College of Medicine).
Des signaux attracteurs tels que la nétrine-1 (via le récepteur DCC), le BDNF (via le récepteur TrkB) et le NGF (via le récepteur TrkA) induisent la synthèse locale de composants cytosquelettiques tels que la β-actine dans les cones de croissance des axones (Leung et al. 2006, Willis et al. 2007, Yao et al. 2006).
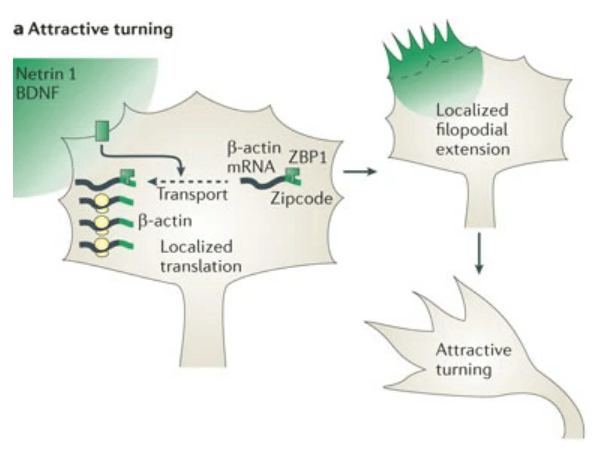
Un commentaire plus détaillé de cette figure est disponible en vidéo.
Source : https://www.nature.com/articles/nrn3210
Les microARN et l’inhibition de la traduction
De curiosité au début des années 2000, les microARN sont passés au statut de composants fréquents et indispensables pour comprendre l’expression des gènes par régulation de la traduction (Bartel, 2018). On estime que près de la moitié des ARNm codés par le génome humain ont un microARN qui les contrôle.
Les microARN (miARN), qui font entre 20 et 25 nucléotides sont produits à partir de transcrits plus longs synthétisés par l’ARN polymérase II, la même qui synthétise les ARNm. Les transcrits primaires sont longs de plusieurs centaines à plusieurs milliers de nucléotides et sont appelés pri (pour primary transcript)-miARN. Un pri-miARN peut contenir la séquence d’un ou de plusieurs miARN. Les miARN sont également produits à partir de certains introns excisés et des régions 3′ non traduites de certains pré-ARNm. Des structures en épingle à cheveux de 70 nucléotides de longueur avec un appariement de bases imparfait dans la tige se forment dans les pri-miARN. Une RNase nucléaire spécifique d’ARN double brin appelée Drosha forme alors dans le noyau un complexe appelé Microprocessor avec une protéine appelée DGCR8 chez l’homme (Pacha chez la drosophile) et clive la région en épingle à cheveux du long précurseur, générant un pré-miARN.
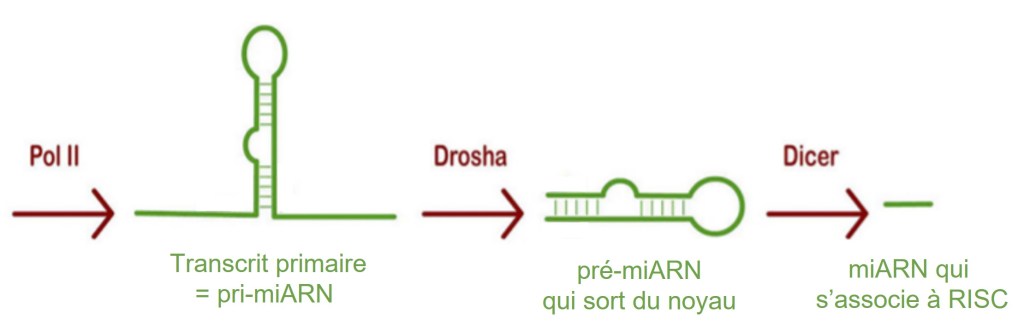
D’après https://www.jbc.org/article/S0021-9258(20)62316-X/fulltext
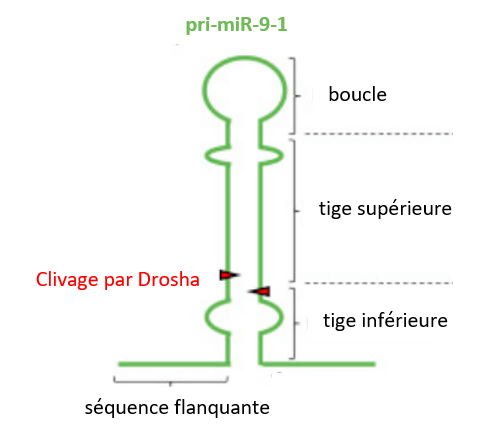
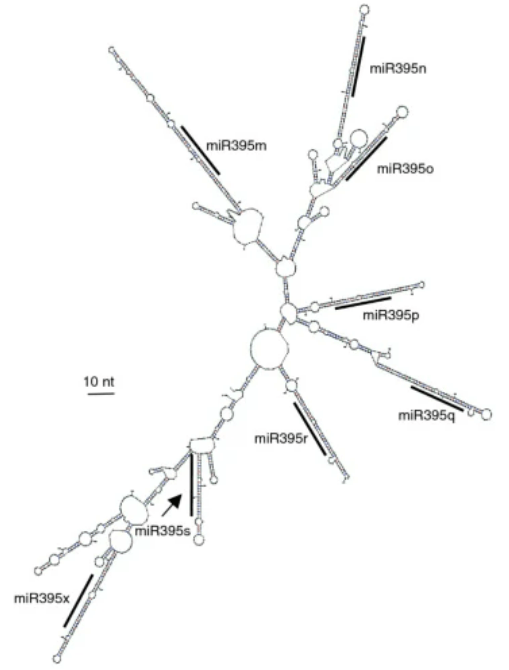
Selon la structure secondaire du pri-miRNA des clivages alternatifs peuvent être produits par Drosha, augmentant le répertoire de cibles dont la traduction sera inhibée à partir d’un seul pri-miRNA (Bofill-De Ros et al., 2019). Certains pri-miRNA sont polycistroniques, c’est-à-dire qu’ils forment plusieurs boucles qui donneront naissance à plusieurs microARN différents (tels miR156, miR166 ou miR395 chez les Angiospermes) (Merchan et al., 2009).
Signalons que des précurseurs de microARN sont aussi générés à partir d’introns selon des modalités différentes de celles décrites ci-dessus. On appelle ces microARN des mirtrons (Ruby et al., 2007).
Quel que soit les modalités de leur production, les pré-miARN sont reconnus et exporté du noyau par l’Exportine-5 (Yi et al., 2003) (l’orthologue chez les plantes s’appelle HASTY (Park et al., 2005)). Une fois dans le cytoplasme, la RNase spécifique de l’ARN appelée Dicer agit avec une protéine de liaison à l’ARN double brin appelée TRBP chez les humains (Loquacious chez la drosophile) pour cliver davantage le pré-miARN en un miARN double brin. Celui-ci fait environ deux tours d’une hélice d’ARN de forme A, avec des brins de 21-23 nucléotides de long et deux nucléotides non appariés à chaque extrémité. Enfin, l’un des deux brins est sélectionné pour l’assemblage dans un complexe de silencing (RISC) où la région 5′ du miARN monocaténaire est attachée à la protéine Argonaute (Iwasaki et al., 2010). Ce complexe Argonaute/miARN est généralement très stable, avec une demi-vie pouvant aller jusqu’à plusieurs jours (Gantier et al., 2011). Le miARN reconnait sa séquence cible, le plus souvent dans la région 3’UTR de l’ARNm cible. Le blocage de la traduction se fait alors de différentes manières, par blocage de l’initiation ou de l’élongation de la traduction. Les protéines de la famille Argonaute sont capables de se lier à la guanosine méthylée de la coiffe 5′ des ARNm et sont essentiels dans ce processus (Djuranovic et al., 2011). Certains miARN et complexes associés sont capables de recruter des endonucléases qui détruisent les ARNm.
Les miARN sont clairement essentiels pour le développement embryonnaire et ce, dès les stades précoces. En effet, le knock-out de Dicer chez la souris aboutit à une impossibilité d’obtenir des miARN matures et les embryons arrêtent leur développement autour de la gastrulation avec une taille réduite et une absence d’induction du mésoderme (Bernstein et al., 2003). De même chez Arabidopsis, le mutant perte-de-fonction dcl1 (Dcl1 est l’équivalent de Dicer chez les plantes) aboutit à une léthalité embryonnaire (Schauer et al., 2002).
Certains miARN peuvent être produits de manière massive et avoir de multiples cibles : c’est le cas de miR-430 chez le poisson-zèbre. Lors de l’activation de la transcription dans l’embryon (MBT), des centaines d’ARNm maternels perdent leur queue polyA et sont dégradés. Ce processus est provoqué par la fixation sur leur 3’UTR de miR-430 qui est codé par un gène qui a 90 copies dans le génome du poisson-zèbre. Plus tard, miR-430 a un rôle dans la neurogenèse (Takacs et Giraldez, 2016). Inversement, l’expression d’un même ARNm peut être contrôlée par plusieurs miARN (comme par exemple MYB2D contrôlé par miR-828 et miR-858 (Guan et al., 2014)).
Des surexpressions ou des expressions trop faibles de miARN ont été associées à des tumeurs et également une partie des déficiences mentales de la trisomie 21 trouve son origine dans la surexpression de miR-155 qui est codé par un gène sur le chromosome 21 (Nuovo et al., 2018). Des protéines peuvent aussi inhiber la maturation des miARN comme Lin28 qui se fixe sur les pre-miARN de Let7 et empêche les clivages nécessaires par Dicer (Nam et al., 2011). Cette régulation joue un rôle important dans le contrôle des cellules souches et la tumorigénèse.
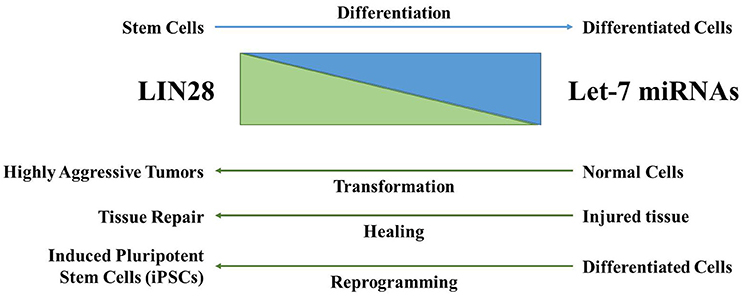
Une version commentée de cette figure est disponible en vidéo.
Source : https://www.frontiersin.org/articles/10.3389/fgene.2017.00031/full
Chez les végétaux, les microARN peuvent être transportés sur de longues distances via les plasmodesmes ou le phloème et agir de manière non-cellulaire autonome. Par exemple, des microARN sont synthétisés dans les tiges en cas de carence nutritive et transportés vers les racines où elles participent à l’activation des réponses pour compenser ces carences (Buhtz et al., 2010, Lin et al., 2008). Chez les Fabacées, miR-2111-3 est un microARN exprimé dans les cellules du phloème des feuilles et qui est transporté par la sève brute et inhibe dans les racines l’expression de TML (TOO MUCH LOVE) qui est un inhibiteur de la formation de nodosités (Tsikou et al., 2018).

LA CARTE MENTALE
QUIZZ
Q1. Associez chaque élément à sa fonction dans la traduction :
- ARNt chargé d’une méthionine
- ARNr 28S
- Codon UAG
- eIF4E
A) Se lie à la coiffe 5′ des ARNm pour initier la traduction
B) Transporte l’acide aminé correspondant au codon d’initiation de la traduction
C) Catalyse la formation de la liaison peptidique
D) Met fin à l’élongation de la protéine
Q2. Vrai ou faux ? Les microARN sont des petits ARN codants qui inhibent la traduction d’ARNm cibles.
Q3. Associez chaque élément au bon rôle dans la biogenèse des microARN :
- Drosha
- Dicer
- Argonaute
- RISC
A) Clive les pri-miARN en pré-miARN dans le noyau
B) Complexe qui associe les microARN matures aux ARNm cibles pour inhiber leur traduction
C) Coupe le pré-miARN en microARN matures dans le cytoplasme
D) Se lie directement aux microARN au sein du complexe d’inhibition de la traduction
Réponses au quizz : Q1. 1B, 2C, 3D, 4A. Q2. Faux car les microARN sont des ARN non codants. Q3. 1A, 2C, 3D, 4B.
VOIR LES EXERCICES SUR L‘EXPRESSION DES GENES
AUTRE RESSOURCE :
LIEN VERS LE GLOSSAIRE DES TERMES LIES A LA GENETIQUE
- Adhérences cellule-cellule
- Arabidopsis thaliana
- Axe antéro-postérieur chez la drosophile
- Biomécanique du développement
- Caenorhabditis elegans
- Concepts principaux
- Contrôle de la transcription
- Contrôle génétique et épigénétique
- Croissance du tube pollinique et double fécondation chez les Angiospermes
- Croissance et guidage axonal
- Des modèles animaux moins classiques
- Développement de l’oeil des Vertébrés
- Développement et évolution
- Et l’Humain ?
- Exercices sur l’ovogenèse, la spermatogenèse et la fécondation
- Exercices sur le contrôle de l’expression des gènes
- Exercices sur le développement des bourgeons de membre
- Exercices sur le développement des muscles striés squelettiques
- Exercices sur le développement des végétaux et les hormones végétales
- Exercices sur les cycles et les divisions cellulaires
- Exercices sur les étapes du développement, les inductions embryonnaires et la mise en place des axes de polarité
- Exercices sur les matrices extracellulaires, le cytosquelette et les adhérences cellule-cellule
- Exercices sur les voies de signalisation
- Glossaire
- Glossaire des termes liés à la génétique
- Glossaire des termes liés au cytosquelette, la matrice extracellulaire, l’adhérence et la migration cellulaire
- Hématopoïèse et développement des cellules du système immunitaire
- Histoire de la biologie cellulaire et de la biologie du développement
- L’acide rétinoïque
- L’apoptose
- L’autophagie
- L’organogenèse
- L’ovogénèse prépare le développement embryonnaire
- La drosophile
- La fécondation
- La formation des somites
- La gastrulation
- La gastrulation (version allégée)
- La métamorphose chez les Hexapodes et les Amphibiens
- La neurogénèse chez les mammifères adultes
- La neurulation
- La poule
- La signalisation calcique
- La souris
- La superfamille TGFβ et ses voies de signalisation
- La voie de signalisation de l’auxine et ses rôles
- La voie de signalisation Hedgehog
- La voie de signalisation Hippo et ses composants YAP/TAZ
- La voie de signalisation Notch
- Le clivage
- Le cytosquelette
- Le destin des cellules et les réseaux de régulation génique
- Le développement des bourgeons de membre
- Le développement des muscles striés squelettiques
- Le développement des organes génitaux et des cellules germinales
- Le développement du cortex
- Le méristème apical caulinaire en phase végétative et lors de la formation d’une fleur
- Le poisson zèbre
- Le xénope
- Les cellules des crêtes neurales
- Les cellules et les gènes en action dans le développement
- Les cellules souches
- Les cellules tumorales
- Les cycles et les divisions cellulaires
- Les étapes du développement
- Les étapes du développement embryonnaire d’Arabidopsis thaliana et leur contrôle
- Les inductions embryonnaires et les gradients de morphogène
- Les matrices extracellulaires animales
- Les organismes modèles
- Les outils pour étudier l’expression et la fonction des gènes
- Les parois des cellules végétales
- Les techniques et les outils pour la biologie cellulaire
- Les transitions épithélio-mésenchymateuses et les migrations cellulaires
- Les vésicules extracellulaires
- Les voies de signalisation
- Les voies de signalisation FGF
- Mise en place des axes chez les Vertébrés
- Structures et processus cellulaires
- Voies de signalisation WNT